 |
|
|  |
|
Die Untersuchung morphologischer (gestaltlicher) Merkmale und die molekulargenetische Analyse (DNA-Sequenzvergleiche) sind zwei voneinander unabhängige Zugänge zur Erfassung von Verwandtschaftsverhältnissen. Es wurde erwartet und wird oft behauptet, dass die Ergebnisse dieser beiden Disziplinen zusammenpassen. Das heißt: Die Datensätze beider Forschungsgebiete sollten dieselben Auffassungen über den Evolutionsweg stützen. Entsprechend wird die Passung von Morphologie und Molekülen als besonders starker Beleg für Evolution gewertet.
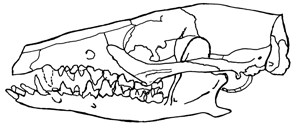 |
| Abb.1: Schädel von Maelestes gobiensis aus der Mongolei. (Nach Wible et al. 2007) |
|
Die Realität ist jedoch vielfach weitaus komplizierter. Die Hoffnung, dass molekular begründete Verwandtschaftsverhältnisse morphologisch und paläontologisch abgeleitete Abstammungsbeziehungen bestätigen würden, hat sich weithin nicht erfüllt. Ein Beispiel dafür liefert die Evolution der Säugetiere. Schon frühere Studien zeigten, dass hier ein Konfliktpotential liegt (Kumar & Hedges 1998). Nun haben ein soeben beschriebenes Fossil und eine neue und gleichzeitig die bislang umfangreichste Auswertung, die auf 409 Merkmalen von 69 fossilen und heutigen Säugetiergattungen basiert, den Konflikt verschärft. Denn nach molekularen Daten sollen die Plazenta-Tiere vor 148 Millionen Jahren entstanden sein, während die morphologisch-paläontologische Analyse für ein rattenartiges Säugetier als ältesten Plazentalier spricht, das auf 63 Millionen Jahre datiert wird (Wible et al. 2007). Cifelli & Gordon (2007, 919) kommentieren: Die jüngsten Untersuchungen repräsentierten den „neuesten Volleyschuss“ in der „Moleküle-versus-Morphologie-Debatte“. Die Ergebnisse verkleinern nicht wie erhofft den Graben zwischen den Schlussfolgerungen der beiden Disziplinen, sondern erweitern ihn. Die größer gewordene Diskrepanz könnte auf zweierlei Weise aufgelöst werden. Zum einen könnten neue Fossilfunde das Bild verändern. Cifelli & Gordon (2007, 919) weisen jedoch auf Studien hin, wonach der Fossilbericht bei den Säugetieren als ziemlich vollständig betrachtet werden kann. Zum anderen könnten die Annahmen über den Gang der molekularen Uhr falsch sein. Immerhin passen die Geometrien der Verwandtschaftsbeziehungen grob zusammen (die Positionen der Afrotheria und Xenarthra bilden jedoch Ausnahmen [Wible et al. 2007, 1005]). Ob sich der gegenwärtige Widerspruch auflösen lässt, wird sich zeigen müssen.
Die Analyse von Wible et al. (2007) erbrachte noch weitere interessante Ergebnisse. Das von ihnen neu beschriebene Fossil Maelestes gobiensis aus der Mongolei (auf 71-75 Millionen Jahre datiert) und sämtliche anderen Säugergattungen aus der Kreide müssen auf tote Seitenäste des hypothetischen Stammbaums gestellt werden. Zu Beginn des Tertiärs tauchen dann die meisten „modernen“ Säugergruppen in der Fossilüberlieferung explosionsartig auf. Cifelli & Gordon (2007) ziehen einen Vergleich mit der kambrischen Explosion. Während man aus der Kreide nur 40 fossile Säugetier-Gattungen kennt, sind es im Tertiär über 4000. Die verwandtschaftlichen Beziehungen der Kreide-Gattungen zu den modernen Plazentaliern sind durchweg stark umstritten (Wible et al. 2007, 1003). Fazit: Einmal mehr führt die Zunahme an Kenntnissen nicht zu größerer Klarheit in Ursprungs- und Abstammungsfragen.
[Cifelli RL & Gordon CL (2007) Re-crowning mammals. Nature 447, 918-920; Kumar S & Hedges BS (1998) A molecular time-scale for vertebrate evolution. Nature 392, 917-920; Wible JR, Rougier GW, Novacek MJ & Asher RJ (2007) Cretaceous eutherians and Laurasian origin for placental mammals near the K/T boundary. Nature 447, 1003-1006]
|

|
Für die Entstehung homochiraler Bausteine für die Synthese von Biomakromolekülen liegt nach wie vor keine befriedigende rein kausalmechanistische Erklärung vor (Imming 2006). Wie können Moleküle, die in spiegelbildlichen Strukturvarianten (Enantiomeren; vgl. Abb. 1) vorkommen und sich in ihren chemischen Eigenschaften praktisch nicht unterscheiden (wie z.B. Aminosäuren und Kohlehydrate) so getrennt werden, dass daraus einheitliche Makromoleküle aufgebaut werden können?
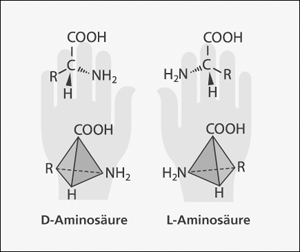 |
| Abb.1: D- und L-Aminosäure. Die beiden Formen sind spiegelbildlich zueinander. Alle Aminosäuren haben die gleiche Grundstruktur (L-Aminosäuren) und unterscheiden sich nur im Rest R. In Ursuppenexperimenten werden immer beide Formen zu gleichenTeilen gebildet (Razemate). (Aus Junker & Scherer 2006) |
|
Ohne homochirale Bausteine ist Leben – wie wir es kennen – nicht möglich. Die Trennung von Enantiomeren gelingt im Labor, erfordert jedoch zwingend den Einsatz homochiraler Chemikalien sowie geplante und regulierte Versuchsabläufe.
Schon seit längerem und auch jetzt wieder in neuen Untersuchungen sind Techniken ausgearbeitet worden, um Mischungen mit einem geringen Enantiomerenüberschuss so anzureichern, dass anschließend ein Enantiomer stark überwiegt. Perry et al. (2007) haben gefunden, dass das D/L-Gemisch der Aminosäure Serin mit einem geringen Enantiomerenüberschuss durch Sublimation angereichert werden kann. Bei der Sublimation geht ein Stoff direkt aus der festen Phase in die Gasphase über (und umgekehrt). Dieses Phänomen ist thermodynamisch gut beschreibbar und wird im Labor z.B. zur Reinigung von Reaktionsprodukten genutzt. Klussmann et al. (2006) haben kürzlich gezeigt, dass eine Anreicherung von Aminosäure-Enantiomeren auch durch Nutzung des Phasenübergangs fest – gelöst möglich ist.
Die Publikation von Perry et al. (2007) veranlasste Ben Feringa und Mitarbeiter, eigene Untersuchungen über die Anreicherung von Enantiomeren durch Sublimation zu veröffentlichen (Fletscher et al. 2007). Sie sublimierten verschiedene Aminosäuren mit einem geringen Enantiomerenüberschuss von 0,5 bis 10%. Das im Überschuss vorliegende Enantiomer wurde dabei angereichert. Dabei darf nur eine geringe Menge (im Promille-Bereich) des Ausgangsmaterials sublimiert werden. Der Effekt zeigt außerdem eine Abhängigkeit von Druck und Temperatur. In der Überschrift vermitteln die Autoren den Eindruck, einen relevanten astrophysikalischen Mechanismus zur Anreicherung von L-Aminosäuren vorzustellen. Aufgrund der bisher vorliegenden Daten sollte diese Behauptung allerdings kritisch hinterfragt werden. Im Kosmos können lokal hohe Temperaturunterschiede auftreten und es herrscht ein stark reduzierter Druck (Vakuum). Diese Randbedingungen sind für Sublimation günstig, trotzdem wäre zu zeigen, wie gut eine Sublimationsapparatur die Situation im Kosmos modelliert. Nachzuweisen bleibt ebenso, ob der Anreicherungseffekt auch bei Ausgangssituationen auftritt, in denen nicht reine Aminosäuren vorgelegt werden, sondern komplexe Substanzgemische mit Spuren von Aminosäuren, wie das unter präbiotischen Bedingungen zu erwarten ist. Wie soll außerdem unter ungesteuerten Bedingungen sichergestellt werden, dass nur geringste Anteile (Promille) der Aminosäuren sublimiert werden?
Abschließend sei auf die schon vor längerem von Klaus Dose (1987) gemachte Feststellung hingewiesen: „Es gibt zwar schwache asymmetrische Kräfte in der unbelebten Natur, aber jedes geringfügige Vorherrschen von D- oder L-Formen bei präbiotischen Prozessen würde in einer geologischen Umgebung durch Razemisierungsreaktionen wieder aufgehoben werden.“ An dieser Einschätzung hat sich bis heute nichts geändert, wie Cataldo (2005) in ihrem Übersichtsartikel belegen, wenn sie schreiben: „The enantiomeric excess found in meteorites usually involves unusual aminoacids which are not common in terrestrial environment and which are much less prone to racemization than the corresponding terrestrial a-aminoacids which instead are usually found as chiral mixtures.“
Unter natürlichen Bedingungen, z.B. im Kosmos, razemisieren Aminosäuren schneller, als dass durch Mechanismen wie die hier beschriebenen Enantiomerenüberschüsse verstärkt werden könnten.
Selbst wenn die aufgezeigten Schwierigkeiten befriedigend gelöst werden könnten, wäre damit immer noch nicht plausibel erklärt, warum in der Natur fast ausschließlich L-Aminosäuren in Proteine eingebaut werden und D-Zucker in Nukleinsäuren. Manche mutig formulierte Überschriften wünschte man sich mit einem Fragezeichen abgeschlossen.
[Cataldo F, Brucato JR & Keheyan Y (2005) Chirality in prebiotic molecules and the phenomenon of photo- and radioracemization, J. Phys., Conference Series 6, 136-148; Dose K (1987) Präbiotische Evolution und der Ursprung des Lebens. Chemie in unserer Zeit 21, 177-185; Fletcher SP, Jagt RBC & Feringa BL (2007) An astrophysically-relevant mechanism for amino acid enantiomer enrichment. Chem. Commun. 2578-2580; Imming P (2006) Die fehlenden Spiegelbilder. Erklärungsversuche für das Phänomen der natürlichen Homochiralität. Stud. Int. J. 13, 14-21 und http://www.genesisnet.info/schoepfung_evolution/e42081.php; Klussmann M, Iwamura H, Mathew SP, Wells Jr DH, Pandya U, Amstrong A & Blackmond DG (2006) Thermodynamic control of asymmetric amplification in amino acid catalysis. Nature 441, 621-623; Perry RH, Wu C, Nefliu M & Cooks RG (2007) Serine sublimes with spontaneous chiral amplification. Chem. Commun. 1071-1073] |

|
Anfang der 1990er Jahre wurden Aufsehen erregende Berichte über Mikroorganismen in Bernstein aus Süddeutschland veröffentlicht. Nahe dem Schliersee (Bayern) hatte U.-Ch. Bauer, ein Fossiliensammler, in mesozoischen Schichten kleine Bernsteinstücke gefunden und wissenschaftlicher Bearbeitung zugänglich gemacht. Zunächst war das Bernsteinvorkommen den Raibler Schichten (Karn, Obere Trias, entspricht einem radiometrischen Alter von 220-230 Ma) zugeordnet worden (Poinar 1993). Diese Angaben waren in mehreren Publikationen zitiert worden. Schmidt et al. (2001) legten aufgrund eigener Feldarbeit sowie anhand biostratigraphischer und petrographischer Untersuchungen eine andere geologische Zuordnung des Bernsteinvorkommens vor, nämlich Cenoman (Obere Kreide, entspricht einem radiometrischen Alter von 93-99 Ma). Diese neue Einstufung machte auch eine neue Interpretation der im Bernstein enthaltenen fossilen Mikroorganismen notwendig. So sind z.B. vergebene Namen wie Paramecium triassicum und Triasamoeba alpha unpassend für Organismen aus der Kreide.
In einer neuen Arbeit beschreiben nun Schmidt et al. (2006) Bernstein aus den Südlichen Alpen, dessen Vorkommen sie in die Trias stellen. Die millimetergroßen, tropfenförmigen Bernsteinstücke befinden sich in einem Paläoboden (paleosol) in der Nähe der Italienischen Stadt Cortina d’Ampezzo. Dieses Vorkommen ordnen die Autoren der Heiligkreuz/Santa Croce Formation in den Dolomiten zu. Es war bereits zuvor von Roghi et al. (2006) beschrieben worden.
Aufgrund ihrer mikroskopischen Untersuchungen der Bernsteintröpfchen beschreiben und dokumentieren Schmidt und Mitarbeiter eingeschlossene Bakterien (sie stellen die Mehrheit der eingeschlossenen Mikroorganismen), Pilze (darunter solche, die der heutigen Gattung Ramularia ähnlich sind) und Algen. Außerdem konnte unter den Ciliaten (Wimperntierchen) ein Vertreter der modernen Gattung Coleps (Colepidae) identifiziert werden. Mehrere Arten beschalter Amöben (Testacea) aus den Familien Centrooyxidae und Diffugidae wurden nachgewiesen, wobei dies derzeit deren frühester Nachweis für nichtmarine Testacea ist.
Unter ökologischen Gesichtspunkten dokumentieren diese triassischen Bernsteineinschlüsse alle Ebenen einer Mikrobiozönose, also eines kleinen Lebensraums: Bakterien und phototrophe Algen (Produzenten), Protozoen (Verbraucher) und Pilze als (Zersetzer). Aufgrund der Mikrobenzusammensetzung in einzelnen Bernsteintröpfchen vermuten die Autoren, dass sie nicht am Boden, sondern an nassen Stellen auf den harzproduzierenden Pflanzen gelebt haben.
Schmidt et al. bemerken, dass die beschriebenen Einschlüsse Einblicke in die Evolution und Paläo-Ökologie von Mikroorganismen aus dem unteren Mesozoikum liefern. Dabei hat es den Anschein, als habe sich auf der untersten Ebene der Nahrungskette seit der Trias morphologisch nichts oder nur wenig verändert. Abschließend unterstreichen die Autoren, dass aufgrund ihrer Befunde Gattungen und sogar Arten von Mikroorganismen in der Lage waren, geologische Epochen zu überleben. Weiter oben hat sich die Nahrungskette aufgrund veränderter Umweltbedingungen (z.B. Massenaussterben an der Grenze Kreide-Tertiär) gewandelt. Seit dem unteren Mesozoikum haben die Protozoen unverändert die Zeit der Dinosaurier wie auch die Veränderungen unter den Angiospermen (bedecktsamigen Blütenpflanzen), Vögeln und Säugetieren überlebt. Man darf gespannt sein auf Erklärungsversuche für die unterschiedliche Auswirkung von Selektionsdrücken.
[Poinar GO, Jr, Waggoner BM & Bauer U-C (1993) Terrestrial soft-bodied protists and other micro-organisms in Triassic amber. Science 259, 222-224; Roghi G, Ragazzi E & Gianolla P (2006) Triassic amber of the Southern Alps (Italy). Palaios 21, 143-154; Schmidt AR, Eynatten Hv & Wagreich M (2001) The Mesozoic amber of Schliersee (southern Germany) is Cretaceous in age. Cretaceous Res. 22, 423-428; Schmidt AR, Ragazzi E, Coppellotti O & Roghi G (2006) A microworld in Triasic amber. Nature 444, 835.]
|

|
Viele Lebewesen können unterschiedliche Gestalten ausprägen, je nachdem welche Umwelteinflüsse auf sie während ihrer Ontogenese (individuelle Entwicklung) einwirken. Ein bekanntes Beispiel ist das Landkärtchen, ein Falter, der deutlich verschiedene Sommer- und Herbstformen ausbildet. Solche Gestaltsvariationen, die nicht auf Unterschiede im Erbgut, sondern auf verschiedene Umwelteinflüsse zurückzuführen sind, nennen die Biologen Polyphenismus („Vielgestaltigkeit“).
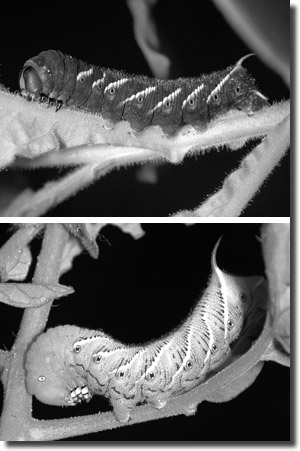 |
| Abb.1: Die schwarze und grüne Form des Tabakschwärmers (Manduca sexta). (Fotos: Yuichiro Suzuki, Abdruck mit freundlicher Genehmigung) |
|
Über die evolutive Entstehung von Polyphenismus und mithin von Umweltsensitivität ist wenig bekannt. Das Problem dabei: „Es benötigt den Erwerb von komplizierten genetischen Kontrollelementen, die die Umweltbedingungen messen und selektiv verschiedene Gengruppen, abhängig von den Bedingungen aktiviert“ (Myers 2006). In einer 2006 veröffentlichten Studie konnten Suzuki & Nijhout unter Laborbedingungen diesen Vorgang experimentell demonstrieren. Der Tomatenschwärmer (Manduca quinquemaculata) zeigt einen Polyphenismus: im kühleren Norden der USA sind die Raupen schwarz, was vorteilhaft für die Absorption von Sonnenlicht und damit Wärme ist. Im wärmeren Süden sind die Raupen dagegen grün und dadurch besser getarnt.
Die Autoren untersuchten nun die Raupe des nahe verwandten Tabakschwärmers (Manduca sexta), die normalerweise immer grün ist. Es existieren aber Mutanten des Tabakschwärmers mit schwarz gefärbten Raupen. Es konnte gezeigt werden, dass die Mutation zu einer Verringerung des Juvenilhormons führt, welches die Färbung der Haut reguliert. Im mutierten Stamm kamen jedoch Raupen vor, die nach einer Hitzeschockbehandlung in einem frühen Entwicklungsstadium unterschiedliche Grünfärbungen aufwiesen. Durch weitere Züchtung mit dieser besonderen Mutante mit sonst schwarzen Raupen erzielten die Wissenschaftler nach nur 13 Generationen einen Stamm, dessen Raupen ab einer bestimmten Temperaturschwelle von 28,5 °C immer grün statt schwarz gefärbt waren. Die höhere Temperatur führte zur Produktion von mehr Juvenilhormon, in dessen Folge wieder die grüne Färbung auftritt. Damit war eine bestimmte Umweltsensitivität (hier Sensitivität auf unterschiedliche Temperatur) und damit die Entstehung eines Polyphenismus experimentell demonstriert, ein Vorgang der auch als genetische Akkomodation bezeichnet wird.
Die Experimente zeigen, dass es – als Vorstufe zur sichtbaren Variation der Färbung – eine unsichtbare Variation gibt, in unserem Fall die Konzentration des Juvenilhormons. Ist diese niedrig, sind die Raupen schwarz gefärbt, ist sie hoch, dann sind sie grün – erst einmal unabhängig von der Temperatur. Die Variation (viel oder wenig Juvenilhormon) kann nun beim gezüchteten Tabakschwärmerstamm bzw. natürlicherweise beim Tomatenschwärmer durch die Temperatur festgelegt und fixiert werden. Es zeigte sich, dass der gezüchtete polyphene Stamm des Tabakschwärmers eine mittlere Konzentration an Juvenilhormon aufweist, die nahe beim Schwellenwert für das Umschalten von „schwarz“ auf „grün“ liegt. Durch die Temperaturerhöhung kann – wie genau ist ungeklärt – die Hormonmenge über den Schwellenwert gehoben werden, mit dem Erfolg, dass die Raupen grün statt schwarz sind (Pennisi 2006). Woher allerdings das ganze System kommt, das die Temperatursensitivität ermöglicht, ist durch diese Versuche nicht gezeigt. Offenbar wurde nur die Regulation des (an sich unsichtbaren) Hormontiters verändert (Suzuki & Nijhout 2006, 652).
Die Tatsache, dass der Tomatenschwärmer natürlicherweise polyphen ist, kann als Hinweis darauf gewertet werden, dass das Potential zur Umweltsensitivität ursprünglich in der Gattung Manduca vorhanden ist und beim Tabakschwärmer teilweise verlorengegangen war, jedoch unter Laborbedingungen wieder reaktiviert werden konnte. Damit wäre auch diese Umweltsensitivität nur ein Ausdruck der Polyvalenz der Stammform. Die Behauptung von Myers (2006), man habe direkt die Evolution eines „komplexen, polygenischen, polyphenen Merkmals“ durch genetische Assimilation und Akkomodation im Labor beobachtet, ist durch die experimentellen Daten nicht gedeckt, wenn damit die de novo-Entstehung gemeint sein sollte.
[Myers PZ (2006) Evolution of a polyphenism. http://scienceblogs.com/pharyngula/2006/02/evolution_of_a_polyphenism.php; Pennisi E (2006) Hidden genetic variation yields caterpillar of a different color. Science 311, 591; Suzuki Y & Nijhout HF (2006) Evolution of a polymorphism by genetic accomodation. Science 311, 650-652]
|

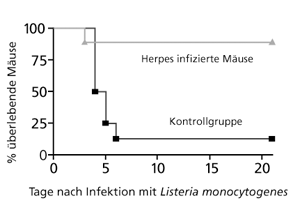 |
| Abb. 1: Mäuse wurden mit einem Herpesvirus infiziert, das in einen latenten Status überging (obere Kurve). Vier Wochen nach der Herpes-Infektion wurde den Mäusen eine Suspension mit dem Krankheitserreger Listeria monocytogenes in den Bauchraum injiziert. Während die mit Herpes infizierten Mäuse fast alle überlebten (obere Kurve), starben fast alle Tiere der Kontrollgruppe (untere Kurve). Nach Barton et al. (2007). |
|
Die Gruppe der Herpesviren besitzt eine doppelsträngige DNA als Erbmaterial. Obwohl alle Menschen im Lauf ihres Lebens vielfache Herpesviren-Infektionen haben, kommt es nur in sehr wenigen Fällen zu einer Reihe von Erkrankungen wie Fieberbläschen, Windpocken, Gürtelrose oder auch Tumore. Nach einer akuten Virusinfektion können Herpesviren ihre Erbsubstanz in die Wirts-DNA einfügen und so in einen latenten Status übergehen, der lebenslang andauert (beim Menschen ist z.B. das Eppstein-Barr-Virus dafür bekannt). Dieser latente Status kann durch Reaktivierung des Virus wieder zum Ausbruch einer Krankheit führen.
Normalerweise werden Viren nur als lästige Schädlinge (Schnupfen) bis hin zu tödlichen Pathogenen (HIV, Ebola) betrachtet. Abweichend von diesem Bild berichten Barton und Mitarbeiter über eine positive Wirkung eines Herpesvirusbefalls bei Mäusen. Wenn Mäuse mit einem Herpesvirus latent infiziert wurden, zeigten sie eine überraschende Resistenz gegenüber den Krankheitserregern Listeria monocytogenes (vor allem für immungeschwächte Personen und schwangere Frauen gefährlich) und Yersinia pestis, dem Pesterreger. Wenn solche Mäuse mit den genannten bakteriellen Krankheitserregern infiziert wurden, überlebten fast alle Herpes tragenden Mäuse, während fast alle „gesunden“ Mäuse (also diejenigen ohne Herpes-Infektion) in den Kontrollgruppen starben (Abb. 1). Dieser Schutz dauerte über einige Wochen an, wurde dann aber schwächer. Die Autoren der Studie konnten nachweisen, dass eine latente Herpesinfektion zu einer deutlichen Stimulation der Produktion bestimmter Signalübertragungsmoleküle (Cytokine) und zur Aktivierung von Fresszellen (Makrophagen) des Immunsystems führt. Der stimulierte Teil des Immunsystems wird auch angeborenes Immunsystem genannt, weil er nicht spezifisch gegen bestimmte Erreger wirkt. Das erklärt, warum eine Virus-Infektion gegen strukturell ganz andere bakterielle Erreger schützen kann.
Die Autoren der Studie vermuten, dass solche Wirkungen auch beim Menschen vorkommen. Sie plädieren dafür, Herpesinfektionen deshalb nicht nur negativ zu sehen und spekulieren über eine symbiotische Komponente mit generellen Vorteilen für die basale Immunabwehr von infizierten Personen. Viren können übrigens auch auf verschiedene Weise zur Variabilität des Erbgutes von Organismen beitragen und wären insofern ebenfalls positiv zu wertende Evolutionsfaktoren, die eine Anpassung an neue Umweltbedingungen erleichtern könnten. Ähnlich wie bei Bakterien sind die allermeisten Viren keine Krankheitserreger. Könnten Viren vielleicht generell positive Funktionen für den Wirt haben und nur in Ausnahmefällen zu Krankheitserregern entartet sein?
[Barton et al. (2007) Herpesvirus latency confers symbiotic protection from bacterial infection. Nature 447, 236-329.]
|

|
Das Röntgenbild des Schädels verblüfft nicht nur den kundigen Betrachter: außen klebt eine dünne helle Hirnschicht wie eine Tapete an der Schädelwand und innen – gähnt blanke Leere. Neben dieser feinen Schicht aus Nervenzellen besteht dieser Gehirnschädel zu etwa 90% aus einer mit Liquor (Nervenwasser bzw. Gehirnflüssigkeit) gefüllten Ventrikel (Hirnkammer) ohne ein einziges zusätzliches Neuron. Und das nicht etwa bei einem geistig Behinderten mit hochpathologischem Gehirnschwund, sondern bei einem normalen 44jährigen Familienvater mit normalen sozialen Beziehungen, der als Verwaltungsbeamter tätig ist. Den Ärzten aus dem Marseiller Spital La Timone, allen voran Lionel Feuillet, einem Neurologen aus der Medizinischen Fakultät der Université de la Méditerranée in Marseille, war so etwas noch nie begegnet.
Dieser Zufallsbefund wurde am 20. 7. 2007 im Fachblatt Lancet beschrieben. Aufgrund einer Lähmungserscheinung im linken Bein wurden Kernspin- bzw Computertomogramme aufgenommen. Als Baby drohte dem Mann ein Hydrocephalus (Wasserkopf), ein relativ häufiges (1 von 1000) pathologisches Geschehen, das in unterschiedlichen Ausprägungen vorkommt. Normalerweise zirkuliert der Liquor und läuft über einen Drainagekanal ab. Bei diesem Patient war er jedoch verengt, was den Abfluss stark erschwerte. Damit der Überdruck nicht weiterhin das motorische Zentrum beeinträchtigte, wurde der überschüssige Liquor mittels einer implantierten Drainage („Shunt“) abgeleitet, bis die Beschwerden verschwanden. Auch beim letzten Eingriff führte die Erneuerung der Drainagenimplantation zum Abklingen der Symptome. Den ersten Eingriff dieser Art erhielt er mit 6 Monaten, den zweiten mit 14.
Das Gehirn selbst wurde durch die mit Liquor gefüllte Hirnkammer extrem stark an den Rand gedrängt. Es hat offensichtlich den verbleibenden peripheren Schädelraum jedoch so optimal genutzt, dass alle vitalen Bereiche ausreichend versorgt wurden. Hier ist natürlich von Bedeutung, dass das Gehirn über einen längeren Entwicklungszeitraum, durch eine langsam erfolgende Verschlechterung die Gelegenheit hatte, Funktionen anders bzw. neu zu verteilen, wobei man auch an „Doppelbelegungen“ denken kann. Das zeugt von einer enormen Plastizität in den neuronalen Verschaltungsmöglichkeiten. Der IQ dieses Mannes liegt zwar mit 75 im untersten Bereich der Normalverteilung (100 ist definitionsgemäß Durchschnitt), doch konnten keine auffälligen Beeinträchtigungen festgestellt werden.
Wissenschaftler stehen nach vielen Jahrzehnten Hirnforschung immer noch vor vielen ungelösten Fragen, wie ein Gehirn funktioniert: ein kleines Detail kann das ganze Gehirn lahm legen und eine große Raumforderung, die das Gehirn auf 10% zusammenschrumpfen lässt, hat nur einen vergleichsweise geringen sichtbaren Effekt. Sicher ist nur, dass Neuronendichte und Verschaltung wichtiger sind als Masse.
Allerdings lässt sich aus dem Befund noch etwas für die Paläanthropologie und damit für die Bewertung fossiler Gehirne ableiten: Hier wird eindrucksvoll bestätigt, dass die Gehirngröße weniger Aussagekraft hat, als dies in Lehrbüchern nahegelegt wird. Von dieser Perspektive sollten Differenzen in den Gehirnvolumina bei fossilen Menschen und möglichen Vorformen nicht allzu viel Bedeutung zukommen – eine immer wieder zu beobachtende Feilscherei um 50 ccm hin oder her dürfte oft keine Bedeutung haben. Dieser Einzelbefund ist auch interessant hinsichtlich der umstrittenen Deutung des winzigen Hobbit-Hirns (vgl. Hartwig-Scherer 2007): Manche Wissenschaftler vermuteten für die Winzigkeit seines Gesamtschädels als Pathologie einen Microcephalus, wobei es sich im Gegensatz zum Hydrocephalus mit äußerlich eher zu großer Schädelkapsel um eine primäre oder sekundäre Wachstumsreduktion des Gehirnschädels handelt bei gleich bleibender Gehirnstruktur (Hartwig-Scherer 2007).
[Feuillet L, Dufou, H & Pelletier J et al. (2007) Brain of a white-collar worker. Lancet 370, 262; Hartwig-Scherer S (2007) Hickhack um den „Hobbit“. Stud. Int. J. 14, 35-37; http://www.nature.com/news/2007/070716/full/070716-15.html]
|

|
Von allen Tierstämmen setzt sich lediglich der Stamm Porifera – die Schwämme – bereits auf einer sehr grundlegenden Ebene ab: Den Vertretern dieses Stamms fehlt es an echtem Gewebe, was ihnen – in Kombination mit ihrem relativ einfachen Grundbauplan – den wenig schmeichelhaften Namen „Nebentiere“ (Parazoa) einbrachte (vgl. Campbell 2007; 754ff.). Zu den gewebelosen Tieren gesellt sich lediglich noch ein weiterer Tierstamm – Placozoa –, der durch eine einzige Art vertreten wird (neue Ergebnisse legen jedoch eine weitaus größere Vielfalt nahe; vgl. Voigt et al. [2004]). Die verbliebene Vielfalt der Tierstämme gehört zur Abteilung (Divisio) der Eumetazoa, die also auch alle Würmer, Gliederfüßer wie Insekten, Fische, Dinosaurier, Vögel und Menschen mit einschließt.
Die älteste Gruppe innerhalb der Eumetazoa, denen von der Evolutionsbiologie ein monophyletischer Ursprung bescheinigt wird, sind die Radiata, die aufgrund ihres zentralen Hohlraums auch als „Hohltiere“ bekannt sind. In ihrem Stamm der Cnidaria finden wir neben Quallen und Korallen auch die Seeanemonen als einer der im Evolutionsparadigma frühesten Vertreter der Gewebetiere überhaupt.
Aufgrund dieser Stellung im evolutionären Gesamtgefüge hatten die Seeanemonen immer als sehr primitive Organismen gegolten – diese aus dem gängigen Evolutionsverständnis abgeleitete Ansicht wurde nun durch eine Genom-Sequenzierung der Sternchenanemone Nematostella vectensis massiv in Frage gestellt. Die Untersuchungen von Putman et al. (2007) legten eine ungeahnte Komplexität offen, die bis dahin nicht für möglich gehalten worden war. Insgesamt, so die Forscher, sei das komplexe Seeanemonen-Genom dem der Wirbeltiere ähnlicher als beispielsweise dem der Fliegen (untersucht an der Fruchtfliege Drosophila melanogaster). Viele der genetischen Eigenschaften der Seeanemone erinnerten gar an das menschliche Erbgut. Diese anfänglich vorhandene und bei den Fliegen und Fadenwürmern im Laufe der vorausgesetzten Evolution wieder verlorene Komplexität widerspricht laut Phillips (2007) dem „weithin vertretenen Standpunkt, dass Organismen durch Evolution komplexer werden“.
Aus der Perspektive der Schöpfungsforschung ist es eine erklärte Erwartung, dass auch geologisch früher auftretende Lebewesen voll entwickelt sind und große Komplexität aufweisen können. Die Befunde von Putnam et al. (2007) passen außerdem zum Konzept des Baukastensystems (Junker 2003), wonach genetische Ähnlichkeiten oberhalb der Grundtyp-Ebene nicht auf eine gemeinsame Stammesgeschichte zurückzuführen, sondern durch einen gemeinsamen Urheber, welcher die selben „Bausteine“ für den Aufbau ansonsten ganz verschiedenartiger Baupläne verwenden kann, während diese Organismengruppen im naturalistischen Evolutionsparadigma weit getrennt sein sollten. Diese Interpretation geht jedoch über eine naturwissenschaftliche Interpretation hinaus und bedarf einer gesonderten Betrachtungsweise.
Dass diese hochkomplexen genetischen Strukturen bereits zu Beginn der Geschichte der Eumetazoa vorhanden sind und nicht erst nach einer langen evolutionären Strecke graduell auftauchen, stellt für gängige Evolutionsvorstellungen eine große Herausforderung dar und passt zu einer Interpretation, welche das Auftauchen neuer Eigenschaften nicht von evolutivem Fortschritt abhängig macht. Interessanterweise ist dieser Eindruck so offensichtlich, dass auch solche Wissenschaftler, welche von einem rein naturgesetzlich erklärbaren Ursprung der biologischen Information durch ungelenkte Prozesse ausgehen, gerne auf „Schöpfungsvokabular“ zurückgreifen – so beispielsweise Daniel Rokhsar, der oben beschriebenes Phänomen folgendermaßen kommentiert: „We have this basic toolkit now for the whole animal kingdom [...] It gives a kind of unity to all animals that I think is kind of surprising“ (Phillips 2007).
Die Frage jedoch, ob diese Einheit durch einen gemeinsamen Vorfahren erreicht wird, oder vielmehr Ausdruck desselben Urhebers ist, ist angesichts oben vorgestellter Forschungsergebnisse offener denn je.
[Campbell NA (2006) Biologie. Pearson Studium. München; Junker R (2003) Baum, Baukasten, Netzwerk. Stud. Int. J. 10; 3-11. Phillips ML (2007) Surprises in sea anemone genome. http://www.the-scientist.com/news/display/53364/; Putnam NH et al. (2007) OrganizationEumetazoan Gene Repertoire and Genomic Sea Anemone Genome Reveals Ancestral. Science 317, 86; Voigt O et al. (2004) Placozoa – no longer a phylum of one. Current Biology 14, R944-945]
|

|
Die Kambrische Explosion stellt nach wie vor eines der größten Rätsel in der Geschichte des Lebens dar. Während einer im konventionellen geologischen Rahmen vergleichsweise kurzen Zeitspanne von nur 10 bis 25 Ma entstanden nahezu alle modernen Tierstämme mit Hartteilen, ohne dass passende Vorfahren bekannt wären. Zusätzlich kommt hinzu, dass die gefundenen Tierstämme über ein komplexes Merkmalsmuster verfügen und miteinander kaum in verwandtschaftliche Beziehungen zu bringen sind (Futuyma 2007; Valentine 2004; vgl. Junker & Scherer 2006). Zur Lösung dieses Problems wurden bereits hypothetische Vorläufer postuliert, die etwa durch geringe Größe, zarten Bau oder schlechte Fossilisationsbedingungen nicht erhalten sein sollen. Die aus dem Präkambrium überlieferten, zum Teil mikroskopisch kleinen Fossilien belegen jedoch, dass eine zarte Struktur nicht prinzipiell einen Hinderungsgrund für eine Fossilisation darstellt (Junker & Scherer 2006, 231). Weiterhin legen Sequenzvergleiche von Genen nahe, dass sich die Entstehung der Tierstämme tatsächlich in kürzester Zeit vollzog, der durch die Fossilien übermittelte Eindruck also die Realität widerspiegelt und kein Trugbild darstellt (Junker 2006). Von der Entdeckung frühkambrischer Fossilien erhofft man sich daher Aufschluss über die Evolution der einzelnen Tierstämme.
 |
| Abb.1: Rippenqualle Mnemiopsis leidyi (Foto und ©: Jan Langmaack; www.tauchen24.info; Abdruck mit freundlicher Genehmigung) |
|
Aus Ningqiang (Süd-China) vermeldet ein internationales Forscherteam den Fund eines auffallend gut erhaltenen Embryos einer Rippenqualle (Ctenophora) aus dem frühen Kambrium (Schopf 2007). Mittels Raman-Spektroskopie und Konfokalmikroskopie konnten bei dem nur 190 Mikrometer messenden Fossil kleinste Strukturen sichtbar gemacht werden, so zum Beispiel die Mundöffnung und die typischen acht Kammreihen, die alle Arten der Ctenophora aufweisen. Auffallend ist allerdings das Fehlen der Tentakel. Die Forscher schließen daraus, dass diese zu einem späteren Zeitpunkt evolviert sind. Der Embryo war von einer Membran umschlossen und befand sich in einem fortgeschrittenen Entwicklungsstadium, kurz vor dem Schlüpfen. Schlüsse auf adulte (erwachsene) Exemplare können gezogen werden, da sich Rippenquallen in allen Entwicklungsstadien ab der befruchteten Eizelle in erster Linie durch ihre Größe unterscheiden, nicht jedoch vom grundsätzlichen Aufbau (Purves 2006, S. 767). Das Alter des Fossils wird mit 540 Ma angegeben (Schopf 2007, 6291) und reicht damit beinahe bis an den Anfang der Kambrischen Explosion zurück, deren Beginn mittlerweile auf 542 Ma (Futuyma 2007) datiert wird. Der Embryo gleicht mit seinen Merkmalen bereits früher gefundenen Fossilien ausgewachsener Tiere der Art Maotianoascus octonarius aus der Chengjiang-Fauna in Süd-China, die auf ein Alter von 530 Ma datiert werden und damit deutlich jünger sind.
Die Evolution der Rippenquallen liegt nach wie vor im Dunkeln, bisher wurden lediglich Vendobionten der Edicara-Fauna als Vorfahren vorgeschlagen. Die Autoren merken jedoch an, dass dafür viele undokumentierte Zwischenschritte nötig wären, um die tiefgreifenden Veränderungen in Bau und Funktion zu realisieren. Die Rippenquallen wurden auch schon als Zwischenglied zwischen den Schwämmen und Nesseltieren vermutet, hier treten jedoch noch größere Schwierigkeiten beim Versuch auf, diese Stämme miteinander zu verbinden (Schopf 2007, 6291).
Mit dem Fund dieser ältesten bekannten Rippenqualle konnten keine neuen Hinweise auf eventuelle Vorfahren oder die Verwandtschaftsbeziehungen zu anderen Tierstämmen gewonnen werden. Vielmehr wurde ein weiteres Mal bestätigt, dass bereits beim erstmaligen Auftauchen der einzelnen Tierstämme im Kambrium differenzierte Baupläne vorlagen, die deutlich voneinander abgegrenzt sind und zum Teil über viele „hochentwickelte“ Merkmale verfügen. Wieder einmal wird deutlich, dass mehr „Wissen“ nicht unbedingt mehr Klarheit für naturalistische Ursprungsmodelle bedeutet – die Lage hat sich im Gegenteil verschärft.
[Futuyma DJ (2007) Evolution. München, 97-99; Junker R (2006) Entstehung der kambrischen Tierwelt – auch nach molekularen Daten explosiv. Stud. Int. J. 13, 46f.; Junker R & Scherer (2006) Evolution – ein kritisches Lehrbuch. Gießen, 229 ff.; Purves WK (2006) Biologie. München, 766 f.; Schopf WJ (2007) Raman spectra of a Lower Cambrian ctenophore embryo from southwestern Shaanxi, China. Proc. Natl. Acad. Sci. 104, 6289-6292; Valentine JS (2004) On the origin of phyla. Chicago und London.]
|

|
Das Tierreich besteht aus zwei großen Untergruppen, den zweiseitig symmetrischen Tieren (z.B. Wirbeltiere) und den radiärsymmetrischen, wie Schwämme, Quallen und Rippenquallen. Ein merkwürdiger, kleiner Wurm (Buddenbrockia), der als Parasit in Moostierchen (Bryozoa) lebt, entzog sich lange Zeit einer genauen Einordnung. Das kleine Tier ist wurmförmig und führt auch wurmartig-schlagende und schnelle Bewegungen aus. Es besitzt keinen Magen, keine inneren Organe oder Sinnesorgane. Entlang der Körperachse befinden sich unter der Haut vier Muskelstränge, mit denen dieses Tier seine wurmartigen Bewegungen ausführt. Diese Parasiten bilden Sporen, die sich an neue Wirte durch nesselzellähnliche Kapseln anheften. Dieser letztere Befund legte für manche Forscher bereits eine Einordnung in der Nähe der Nesseltiere (Cnidaria), zu denen die Quallen gehören, nahe. Dagegen sprach jedoch die eigentümliche Art der Bewegung, die bei den Nesseltieren so nicht vorkommt, eher für eine Einordnung innerhalb der zweiseitig symmetrischen Tiere, z.B. in der Nähe der Fadenwürmer (Nematoden).
Eine neue genetische Analyse plazierte diesen Wurm, wenn auch nur mit Mühe, in die Nähe der Medusen (Quallen). Laut den Wissenschaftlern wurde hiermit zum ersten Mal ein eindeutig radiärsymmetrisches Tier beschrieben, welches sich wie ein typisch zweiseitig symmetrisches bewegt und auch so aussieht. Jiménez-Guri und Mitarbeiter (2007) vermuten, dass sich der wurmförmige Körper innerhalb der Nesseltiere unabhängig von anderen Würmern in einer parallelen Evolution herausgebildet habe und schreiben (Übertragung): „Parallele Evolution, die zur Entstehung des wurmförmigen Körpers führte, könnte ein konserviertes Entwicklungssystem der Musterbildung […] zwischen den zweiseitig symmetrischen Tieren und den Nesseltieren ausgenutzt haben.“ Die Mechanismenfrage, wie eine solche komplexe Parallelbildung entstehen kann, bleibt wohl zukünftiger Forschung vorbehalten.
[Jiménez-Guri E, Philippe H, Okamura B & Holland PWH (2007) Buddenbrockia is a cnidarian worm. Science 317, 116-118.]
|

|
Angesichts der Bedeutung von Nukleinsäuren für lebende Zellen spielen diese Biopolymere auch in der Diskussion über mögliche Szenarien zur Lebensentstehung eine prominente Rolle (z. B. RNA-Welt). Dabei fällt auf, dass auch nach über 80 Jahren experimenteller Untersuchungen – wenn man Oparins Publikation von 1924 als Ausgangspunkt nimmt – noch keine Synthesen für die molekularen Bausteine einer Zelle vorgestellt wurden, von denen man annehmen kann, dass sie zufällig, ohne speziell herbeigeführte und aufrecht erhaltene Randbedingungen ablaufen könnten. Diese unbefriedigende Situation stellt für Chemiker, die sich mit der präbiotischen Chemie der Lebensentstehung beschäftigen, eine große Herausforderung dar.
Sutherland und seine Mitarbeiter haben eine Untersuchung vorgelegt, in der sie basierend auf einer Synthese, die Sanchez & Orgel (1970) veröffentlicht haben, eine alternative Synthese eines Ribosederivats (Ribose-2-aminooxazolin) beschreiben (Anastasi et al. 2006). Dabei umgehen sie die Synthese von freier Ribose; für diese Aldopentose liegt bisher keine plausible präbiotische Synthese vor und außerdem ist dieser Zucker chemisch nicht sehr stabil. Das Ribosederivat lässt sich – bei reinen Ausgangsverbindungen (!) – in erstaunlich hoher Ausbeute (44%) aus 2-Aminooxazolin und Glycerinaldehyd in wässriger Lösung gewinnen. Ribose-2-aminooxazolin ist unter den Reaktionsprodukten das am wenigsten lösliche, so dass das gewünschte Produkt durch Kristallisation gewonnen wurde. Liegt eines der beiden Enantiomeren in einem Überschuss von 60% vor, so erhalten die Autoren durch Kristallisation enantiomerenreines L- bzw. D-Ribose-2-aminooxazolin.
Durch Umsetzung von Ribose-2-aminooxazolin mit Cyanoacetylen kann man a-D-Cytidin in einer Ausbeute von 10-20% erhalten.
Nukleoside in Nukleinsäuren sind b-konfiguriert (der N-Heterocyclus ist in der ebenen Darstellung nach oben angeordet). Also muss a-D-Cytidin (Anordnung der N-Heterocyclus nach unten) in die b-Verbindung umgewandelt werden. Diese Anomerisierung – Konfigurationsumkehr an einem asymmetrisch substituierten Kohlenstoffatom – kann durch Bestrahlung herbeigeführt werden. Die Ausbeute an dem gewünschten Produkt ist allerdings sehr gering (4-6%). In einer jüngsten Studie untersuchten Sutherland et al. diese Umwandlungsreaktion mit dem Ziel, den Mechanismus zu verstehen und die Reaktion hinsichtlich der Ausbeute zu optimieren (Powner et al. 2007). Darüber hinaus wurde noch die Einführung von Phosphatgruppen untersucht. Sie werden ebenfalls zum Aufbau von Nukleinsäuren benötigt.
1H-NMR Untersuchungen lieferten Hinweise dafür, dass unter dem Einfluss der Bestrahlung die Nukleobase zerstört wird und dies ebenso wie die Abspaltung des N-Heterocyclus von der Ribose Ursachen für die geringen Ausbeuten sind. Bei der Bestrahlung von a-D-Cytidin-5’-phosphat erhielten Powner et al. (2007) das gewünschte b-Produkt in 16% Ausbeute (Sanchez & Orgel [1970]: 6%). Umfangreiche 1H-NMR Studien zeigten auch hier, dass durch die Bestrahlung die Ausgangsverbindung abgebaut und umgelagert wird, was zu starkem Ausbeuteverlust führt. Für Cytidin-3’-phosphat ist ebenfalls ein weitgehender Abbau und damit eine geringe Ausbeute zu erwarten. Die Autoren kündigen weitere Studien mit Cytidin-2’-phosphat an, bei dem die beobachteten Abbaureaktionen nicht erwartet werden.
Nach bisherigen Erkenntnissen lassen sich also Cytosin- und Cytidinverbindungen in erstaunlich guter Ausbeute unter Bedingungen synthetisieren, die als präbiotisch gelten könnten, d.h. ohne äußerst hypothetische Randbedingungen. Es entstehen dabei allerdings die falschen Raumstrukturen (a- anstatt der b-Verknüpfung). Die Resultate von Sutherland et al. zeigen, dass die Umwandlung durch Bestrahlung den größten Teil der Vorprodukte wieder zerstört. Außerdem liegen für das eingesetzte 2-Aminooxazolin derzeit noch keine Synthesen vor, die unter präbiotischen Bedingungen plausibel sind. Ein aus dem Blickwinkel der Chemie zur Lebensentstehung verheißungsvoller Weg scheint sich wieder einmal als Sackgasse zu entpuppen in dem Sinne, dass zwar eine gute Idee für eine Synthese eines Bausteines für Biomoleküle angegeben wird, die benötigten Ausgangsstoffe dabei aber vorausgesetzt werden, ohne dass eine plausible Synthese dafür vorgestellt wird. Außerdem wird die Ausbeute im Verlauf der mehrstufigen Synthese marginalisiert. Gespannt darf man auf die weiteren Ergebnisse aus der Arbeitsgruppe von Sutherland warten; werden sie Wege aus dieser Sackgasse aufzeigen können?
[Anastasi C, Crowe MA, Powner MW & Sutherland JD (2006) Direct assembly of nucleoside precursors from two- and three-carbon units. Angew. Chem. 118, 6322-6325; Angew. Chem. Int. Ed. 45, 6176-6179; Powner MW, Anastasi C, Crowe MA, Parkes AL, Raftery J & Sutherland JD (2007) On the prebiotic synthesis of ribonucleosides: photoanomerization of cytosine nucleosides and nucleotides revisited. Chem. BioChem. 8, 1170-1179; Sanches RA & Orgel LE (1970) Studies on prebiotic chemistry: V. Synthesis and photoanomerization of pyrimidin nucleosides. J. Mol. Biol. 47, 531-543.]
|

|
Lebende Zellen besser zu verstehen und zukünftig vielleicht sogar selbst herstellen zu können, ist eine seit vielen Jahren formulierte Herausforderung für die Biowissenschaften. Im Rahmen dieser Bemühungen stehen Projekte, in welchen nach dem kleinstmöglichen Genom für Mikroorganismen gesucht wird oder die denkbar einfachsten Zellen erforscht werden. Unter dem Begriff „synthetische Biologie“ werden diese Bemühungen zusammengefasst (http://www.syntheticbiology3. ethz.ch). Craig Venter war mit Aufsehen erregenden Äußerungen und Beiträgen maßgeblich an der Etablierung dieses Arbeitsbereichs beteiligt. Nun wurde aus dem von ihm gegründeten und nach seinem Namen benannten Institut (J. Craig Venter Institute, Rockville, USA) eine Methode veröffentlicht, mit der es erstmals gelungen ist, ein komplettes Genom als freie DNA von einem Mikroorganismus in einen anderen zu übertragen. Venter und seine Mitarbeiter schlagen für diese Methode den Begriff „Genom -Transplantation“ vor (Lartigue et al. 2007). Dabei wird das ursprüngliche Genom der Empfängerzelle komplett durch das neue Genom ersetzt und es prägt Geno- und Phänotyp der veränderten Zelle.
Lartigue et al. benutzten für ihre Arbeiten zwei schnell wachsende und eng verwandte Mycoplasma-Arten (M. capricolum und M. mycoides), wobei M. mycoides als Genomspender und M. capricolum als Empfänger fungierten; umgekehrt funktioniert der Transfer nicht. 76,4% des 1 083 241 bp (bp = Basenpaare) umfassenden Genoms des Spenderstammes deckt sich mit dem kleineren Genom des Empfängerstammes (1 010 023 bp) zu 91,5%, beim Rest (23,6%) handelt es sich um insertierte (eingefügte) Sequenzen, die in M. capricolum nicht vorkommen (beide Genome wurden im Rahmen dieser Arbeit nach der von Venter entwickelten „Schrotschussmethode“ sequenziert).
Im transplantierten Genom aus M. mycoides waren aus methodischen Gründen Gene für Tetracyclinresistenz (tetM) (Antibiotikaresistenz) und b-Galaktosidase (lacZ) (Erzeugung eines blauen Farbstoffs) enthalten. Für die Handhabung des mechanisch gegen Scherkräfte in Pufferlösungen hochempfindlichen DNA-Makromoleküls mussten neue Methoden entwickelt werden. Die geernteten M. mycoides-Zellen wurden in Agarosegel eingebettet und nach Auflösung der Zellen die DNA in den entstandenen Kavernen gereinigt. Durch Gelelektrophorese wurde nachgewiesen, dass für die weiteren Schritte ein vollständiges, ringförmig geschlossenes Bakterienchromosom zur Verfügung stand.
Das so gewonnene Genom wurde mittels der Ethylenglykol-Methode in M. capricolum transferiert. Die so behandelten Zellen wurden in Gegenwart von Antibiotika kultiviert. Nach drei Tagen waren große blaue Kolonien erkennbar (M. mycoides) und nach 10 Tagen kleine blaue und farblose M. capricolum-Kolonien. Neben der Transplantation des Genoms fand offensichtlich auch Rekombination statt. Durch Kontrollexperimente wurde sichergestellt, dass es sich bei den M. mycoides-Kolonien nicht um Infektionen mit ursprünglichen Zellen handelte. Einzelne M. mycoides-Kolonien wurden ausgelesen und unter erhöhter Tetracyclin-Konzentration weiter kultiviert.
Um zu belegen, dass es sich bei den M. mycoides-Kolonien wirklich um Zellen mit transplantiertem Genom handelt, und um Rekombination in diesen Zellen auszuschließen, wurden an den weiterkultivierten Kolonien sowohl der Genotyp (durch Sequenzierung) als auch der Phänotyp (durch Proteom-Analyse und Tests mit Antikörpern, die jeweils spezifisch an die Zelloberflächen von M. mycoides bzw. M. capricolum binden) ermittelt.Nach Optimierung des Verfahrens gelang laut Angaben der Autoren die Genom-Transplantation bei einer Empfängerzelle unter 150 000, wobei der Mechanismus noch nicht verstanden ist.
Damit ist zum ersten Mal ein Transfer des gesamten Genoms zwischen zwei sehr ähnlichen Organismen herbeigeführt und dokumentiert worden. Sollten sich diese Methoden als zuverlässig erweisen, dann würde nach Auffassung von Lartigue et al. (2007) in den Empfängerzellen eine Plattform zur Verfügung stehen, um durch Genom-Transplantation neue Arten mit wählbaren Eigenschaften herzustellen.
Derzeit ist freilich noch unklar, ob die Methode auf andere Organismen übertragbar ist und ob damit auch Genome, die stärker vom Empfängergenom abweichen, erfolgreich transplantiert werden können (Mycoplasma hat im Gegensatz zu den meisten Bakterien keine Zellwand, welche den Transfer von DNA erheblich erschwert). In der Natur kommt die Übertragung von großen Teilen eines Bakteriengenoms von einer Donor- in eine Empfängerzelle im Rahmen der parasexuellen Konjugation vor. Dabei kommt es auch zum Transfer zwischen verschiedenen Arten, sogar zwischen Gattungen. Die Übertragung eines kompletten Genoms auf diesem Wege ist zwar sehr unwahrscheinlich, aber durchaus nicht ausgeschlossen. Die Autoren der neuen Studie haben damit einen komplexen, natürlich vorkommenden Vorgang durch ein biotechnologisches Verfahren ersetzt und damit handhabbar gemacht.
Während die von Lartigue et al. (2007) vorgestellte Methode von manchen als wichtiger Schritt auf dem Weg zu künstlichen Zellen mit minimalem Genom gefeiert wird, mahnen andere Wissenschaftler Überlegungen zu sozialen, ethischen und Sicherheitsaspekten an (Pennisi 2007).
Jedenfalls liegt hier ein weiteres Beispiel dafür vor, dass gegenwärtig ein biotechnologischer Handlungsspielraum verfügbar ist, dem das biologische Verständnis weit hinterherhinkt. Auch machen die Bemühungen der Autoren um weitgehende patentrechtliche Absicherungen nachdenklich im Blick auf die wirtschaftliche Motivation für das Projekt. Inwieweit diese Methoden wirklich dazu beitragen, dass eines Tages künstliche Zellen im Labor erzeugt werden, die nicht mehr von lebenden Zellen unterschieden werden können, bleibt abzuwarten.
[Lartigue C, Glass JI, Alperovich N, Pieper R, Parmar PP, Clyde A., Hutchison III CA, Smith HO & Venter JC (2007) Genome transplantation in bacteria: changing one species to another. Science 317, 632-638; Pennisi E (2007) replacement genome gives microbe new identity. Science 316, 1827.]
|
|  |