 |
Kleinstes Genom einer photosynthetischen
Bakterienzelle als Hinweis auf einen
genetisch „komplexen“ Vorfahren?
von Siegfried Scherer
Studium Integrale Journal
11. Jahrgang / Heft 1 - Mai 2004
Seite 29 - 31
|
 |
Zusammenfassung: Fast gleichzeitig wurden die kompletten Genomsequenzen von vier photosynthetischen Cyanobakterien und Prochlorobakterien veröffentlicht. Diese winzigen Organismen sind ökologisch außerordentlich erfolgreich, weil sie in extrem hoher Anzahl im Meer vorkommen und für einen wesentlichen Teil der globalen photosynthetischen Bindung von Kohlenstoff verantwortlich sind. Die Genomsequenzen von zwei der sequenzierten Prochlorobakterien sind außergewöhnlich kurz. Es wird diskutiert, daß sie durch vielfachen Genverlust aus „komplexeren“ Vorfahren hervorgegangen sind.
|  |
|
Einleitung
Sauerstoffentwickelnde Photosynthese wird nicht nur von Pflanzen und Algen betrieben, sondern findet sich auch in zellkernlosen (= prokaryotischen) Zellen, den Bakterien. Zwei derartige Bakteriengruppen sind bisher bekannt: Cyanobakterien, welche zusätzlich zum alleinigen Chlorophyll a auch über blaue bzw. rote Phycobiliproteine verfügen, die bei der Lichtabsorption eine Rolle spielen; sowie Prochlorobakterien, die wie Pflanzen und Algen Chlorophyll a und Chlorophyll b besitzen. Während Cyanobakterien (früher „Blaualgen“ genannt) schon sehr lange bekannt sind, wurden die Prochlorobakterien erst vor etwa 20 Jahren entdeckt. Man hat ursprünglich angenommen, sie stünden den vermuteten gemeinsamen Vorfahren der Pflanzen und Algen näher als die Cyanobakterien, daher die Silbe „pro“ (= „vor-“ oder „ur-“) im Namen. Heute beurteilt man das aufgrund von Gensequenzvergleichen jedoch anders; viele Wissenschaftler betrachten die Prochlorobakterien auch als Untergruppe der Cyanobakterien.
|
|
Ökologische Bedeutung von sehr kleinem photosynthetischem Plankton
Wie man auch erst seit kurzem weiß, spielen diese beiden Bakteriengruppen eine immense ökologische Rolle. In Form von allerkleinstem Phytoplankton, dem Pikoplankton, sind diese Organismen für 20-40% der Primärproduktion (= Bindung von CO2 in organischen Molekülen durch Photosynthese) in den Ozeanen verantwortlich. Darunter befinden sich zum Beispiel die Cyanobakteriengattung Synechococcus und die Prochlorobakteriengattung Prochlorococcus (Abb. 1). Beide hat man bei früheren Analysen der photosynthetischen Organismen des Süßwassers und Meerwassers zuerst gar nicht erfaßt, weil die winzigen Zellen durch die verwendeten Filter nicht zurückgehalten wurden. Doch Prochlorococcus allein tritt mit rund 1 Million Zellen pro Milliliter Meerwasser auf und beide Gattungen sind mit 25% der globalen Fixierung von CO2 (10 Milliarden Tonnen pro Jahr!) für das globale Gleichgewicht von Bindung und Freisetzung von CO2 (globaler Kohlenstoffzyklus) außerordentlich wichtig. Daher existiert ein großes Interesse am Studium von pikoplanktischen Cyanobakterien bzw. Prochlorobakterien.
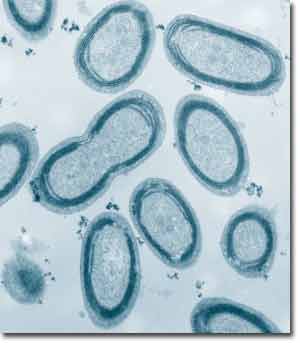 |
Abb. 1: Elektronenmikroskopisches Bild von Prochlorococcus MIT 9313. Die Zellen haben einen Durchmesser von 0,5-0,7 μm. In der Bildmitte ist eine Zelle zu sehen, die sich gerade teilt. Unterhalb der Zellhüllen liegen die als konzentrische dunkle Bänder sichtbaren Thylakoide. Dabei handelt es sich um Membranen, welche den photosynthetischen Apparat tragen (Chlorophyll, Elektronentransportketten u.a.) Abdruck mit freundlicher Genehmigung von Claire Ting und Sallie W. Chisholm, MIT, Cambridge, USA. |
|
|
|
Genomsequenzen
In August 2003 wurden gleich vier Genome von pikoplanktischen Synechococcus und Prochlorococcus veröffentlicht (PALENIK et al. 2003; ROCAP et al. 2003; DUFRESNE et al. 2003, vgl. Tab. 1). Damit sind interessante Genomvergleiche möglich. Wie bei anderen Genomsequenzen von Bakterien ist auch hier etwa ein Drittel der identifizierten, möglicherweise für Proteine codierenden Gene von unbekannter Funktion. Das zeigt, wie wenig wir selbst von Bakteriengenomen verstehen.
Bei allen sequenzierten Arten wurden einzigartige Gene identifiziert, welche jedenfalls teilweise für die Lebensweise als ozeanische Pikoplankter wichtig sein könnten. Die Unterschiede in der chemischen Zusammensetzung des Genoms (GC-Gehalt) der Prochlorococcus-Arten zeigt eine extreme Variabilität; es wird vermutlich nicht bei der Einordnung in eine Gattung bleiben.
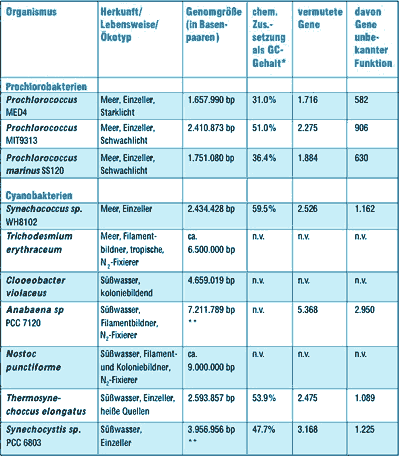 |
Tab. 1: Größe und Zahl der Gene für die zur Zeit bekannten Genome von Pochlorobakterien und Cyanobakterien. Daten aus DUFRESNE et al.(2003), ROCAP et al. (2003) und PALENIC et al. (2003), sowie im Internet unter http://www.sb-roscoff.fr/Phyto/ProSS120/ [letzter Zugriff am 10.09.2003] * GC-Gehalt: Prozentsatz der Nucleobasen Guanin+Cytosin in der DNA. ** In diesen Angaben sind Megaplasmide und Plasmide enthalten. n.v.: Diese Angaben wurden noch nicht veröffentlicht. |
|
|
|
Horizontaler Gentransfer als Evolutionsfaktor
Aus evolutionsbiologischer Sicht sind unter anderem zwei Beobachtungen interessant. Zum ersten wurden für diese Organismen erstmals auch Viren beschrieben (Bakteriophagen). Dazu paßt, daß bei zwei Organismen zahlreiche Sequenzen im Genom gefunden werden, die für eine Einfügung von Fremd-DNA (z.B. von Viren) in das Genom wichtig sind. Diese Sequenzen begrenzen teilweise Genomabschnitte, die einen anderen GC-Gehalt wie das Durchschnittsgenom des betreffenden Organismus enthalten. Das läßt sich gut im Sinne eines vergangenen horizontalen Gentransfers deuten. So könnten diese Organismen nicht nur Gene verloren (s.u.), sondern auch von anderen Arten hinzugewonnen haben. Generell deuten immer mehr Anzeichen darauf hin, daß artübergreifender („horizontaler“) Gentransfer ein wichtiger Variationsfaktor bei mikroevolutiven Veränderungen von Bakterien ist (z.B. GOGARTEN et al. 2002), doch gibt es dazu durchaus auch kritische Stimmen (z.B. KURLAND et al. 2003).
|
|
Genetisch komplexe Vorfahren?
Es fällt auf, daß insbesondere bei Prochlorococcus marinus sehr viele Gene fehlen, die bei anderen photosynthetischen Bakterien vorhanden sind. Beispielsweise vermißt man eine ganze Reihe von Regulationsgenen, mit denen Bakterien sonst auf sich verändernde Umweltreize („Umweltstress“) reagieren. Das wird damit erklärt, daß diese Organismen im Ozean in einer sehr konstanten Umgebung leben, diese Gene nicht mehr benötigen und deshalb im Laufe der Evolution verloren haben. Für einen frei lebenden Organismus, der mit anderen Arten konkurriert, die ein größeres Genom besitzen, ist die Genomgröße von P. marinus ziemlich klein. Die Anpassung an eine ökologisch extrem konstante Schwachlichtzone in 80-200 m Wassertiefe scheint aber als Erklärung noch nicht ganz überzeugend, weil der ebenfalls an Schwachlicht angepaßte Prochlorococcus MIT9313 ein deutlich größeres Genom hat, während der an Starklicht angepaßte Prochlorococcus MED4 sogar ein noch kleineres Genom hat (Tab. 1). Nun sind diese Zellen mit 0,5-0,7 μm Zelldurchmesser und einem Volumen von 0,1μm3 sehr klein. Das hat viele Vorteile, beispielsweise bei der Geschwindigkeit des Stoffaustausches mit der Umgebung oder als Schutz vor dem Verzehr durch Protozoen (das sind zellkernhaltige Einzeller, die sich von Bakterien und Algen ernähren). Könnte mit der Kleinheit des Organismus auch eine grundsätzliche Grenze für die Größe des Genoms gegeben sein? Letztlich scheidet die Zellgröße von Prochlorococcus aber als Erklärung für das kleine Genom aus, weil man mit Pelagibacter ubique eine Zelle mit nur 0,1μm3 Volumen gefunden hat, deren Genom trotzdem etwa 1.500.000 Basenpaare umfaßt (RAPPE et al. 2002).
Genverlust als Evolutionsprozeß ist bereits für verschiedene Krankheitserreger bzw. Symbionten angenommen worden; der bisherige Extremfall ist Mycoplasma genitalium mit einer Genomgröße von 580.000 Basenpaaren mit 468 vermuteten Genen. Dieser ausschließlich parasitische Erreger lebt im Urogenitaltrakt und verursacht dort Entzündungen. DUFRESNE et al. (2003) vermuten für Prochlorococcus ebenfalls einen Genverlust. Dies wird aus DNA-Sequenzvergleichen mit anderen Cyanobakterien geschlossen. Damit stünde auch für diese ökologisch äußerst erfolgreichen, freilebenden Mikroben ein genetisch reichhaltiger ausgestatteter („komplexer“) Vorfahr zur Diskussion, dessen Ursprung jedoch im Dunkeln liegt.
|
|
Die Suche nach dem „minimalen Genset“
DUFRESNE et al. (2003) sprechen davon, daß die Genomgröße von Prochlorococcus sich dem minimal nötigen Genom für ein photosynthetisches Bakterium mit Pflanzentyp-Photosynthese annähert. Diese Deutung erscheint jedoch ein wenig verfrüht – man benötigt für eine solche Aussage sicherlich noch viele Sequenzen vergleichbarer Mikroorganismen sowie funktionale Analysen. Auch dürften die Autoren diese „minimale Genomgröße“ auf eine Situation beziehen, in der die Zelle mit anderen, ähnlich leistungsfähigen Organismen konkurrieren muß. In einer solchen Situation werden sicherlich mehr Gene benötigt als beim Wachstum einer einzelnen Art im Labor unter abgeschirmten Bedingungen. Bisherige Versuche mit anderen Mikroorganismen zur systematischen Zerstörung von Genen (z.B. KOBAYASHI et al. 2003) deuten jedenfalls darauf hin, daß man wohl auch bei Prochlorococcus marinus noch zahlreiche Gene ausschalten kann, ohne die Überlebensfähigkeit der Zellen im Labor zu stören. Welche Konsequenzen solche Genverluste im natürlichen Umfeld haben, vermag derzeit aber niemand zu sagen.
Heute nimmt man aufgrund von kleinsten Genomen parasitisch lebender Bakterien und Geninaktivierungsexperimenten an mehreren Arten an, daß der minimale Genset einer freilebenden Bakterienzelle bei 500-600 Genen liegt (KOONIN 2003), doch für einen minimal komplexen, autotrophen Photosynthetiker muß man vermutlich eine größere Zahl von Genen veranschlagen. Allerdings ergeben sich selbst aus 500 Genen als minimalem Genom für den berühmten LUCA („Last Universal Common Ancestor“) unüberwindlich scheinende Hindernisse für eine kausale Theorie zur Entstehung der ersten Zelle, wenn sich diese Zahl nicht mit plausiblen Argumenten noch deutlich reduzieren läßt.
|
Literatur
- DUFRESNE A et al. (2003)
- Genome sequence of the cyanobacterium Prochlorococcus marinus SS120, a nearly minimal oxyphototrophic genome. Proc. Natl. Acad. Sci. USA 100, 10020-10025.
- FRASER et al. (1995)
- The minimal gene complement of Mycoplasma genitalium. Science 270, 397-403.
- GOGARTEN JP, DOOLITTLE WF & LAWRENCE JG (2002)
- Prokaryotic evolution in light of gene transfer. Mol. Biol. Evol. 19, 2226-2238.
- KOBAYASHI K et al. (2003)
- Essential Bacillus subtilis genes. Proc. Natl. Acad. Sci. USA 100, 4678-4683.
- KURLAND CG, CANBACK G & BERG OG (2003)
- Horizontal gene transfer: a critical review. Proc. Natl. Acad. Sci. USA 100, 9658-9662.
- PALENIK B et al. (2003)
- The genome of the motile marine Synechococcus. Nature 424, 1037-1042.
- RAPPE MS et al. (2002)
- Cultivation of the ubiquitous SAR11 marine bacterioplankton clade. Nature 418, 630-633.
- ROCAP G et al. (2001)
- Genomic divergence in two Prochlorococcus ecotypes reflects oceanic niche differentiation. Nature 424, 1042-1047.
-
-
|
|  |
