 |
Bakterielle Endosymbionten von Pflanzenläusen
mit stark reduzierten Genomen von Siegfried Scherer
Studium Integrale Journal
14. Jahrgang / Heft 2 - Oktober 2007
Seite 66 - 73
|
 |
Zusammenfassung: Die Genomgrößen von DNA-haltigen Organellen (Mitochondrien, Chloroplasten), symbiontisch lebenden Bakterien, Parasiten, Krankheitserregern und frei lebenden Bakterien deuten immer mehr auf ein nahezu lückenloses Kontinuum hin. Dieser Trend hat sich durch einen Bericht über das kleinste bakterielle Genom des Endosymbionten* Carsonella bestätigt, der in spezialisierten Organen von Blattläusen lebt und dessen Genom nur 162 kb* lang ist. Dagegen ist das größte Mitochondriengenom der Tabakpflanze mit 439 kb fast drei Mal so lang. Man vermutet, dass Carsonella von einem frei lebenden g-Proteobakterium abstammt und mehr als 90% seiner Gene verloren hat. Dieses Größenkontinuum ist ein gutes vergleichend-biologisches Argument für die Endosymbiontenhypothese, nach welcher Mitochondrien* und Chloroplasten von frei lebenden Bakterien abstammen. Allerdings kennt man bis heute keinen evolutionären Mechanismus, der eine erstmalige Etablierung von Organellen mit funktionalem Gentransfer (z.B. Mitochondrien) befriedigend erklärt. Es ist derzeit unklar, ob in der Symbiose zwischen Carsonella und dem Blattfloh ein Gentransfer vom Bakterium in den Wirt erfolgt sein könnte. Wenn dazu noch aus dem Wirt Proteine in den Symbionten transportiert würden, was ebenfalls unbekannt ist, dann könnte man Carsonella als ein Organell mit neuartiger Funktion bezeichnen. Das wäre eine aufregende Entdeckung. Obgleich lange bekannt, stellen sich Vergesellschaftungen (Metabiosen, Symbiosen) von Bakterien mit Wirbellosen und Wirbeltieren sowie mit eukaryonten* Mikroben als unerwartet häufig und sehr variabel heraus. Vielleicht erweisen sich Bakterien sowie potentielle Wirte sogargenerell als prädisponiert für funktionale Interaktionen. Es könnte sein, dass im Rahmen solcher Assoziationen vielfältige mikroevolutive Veränderungen möglich sind. So gibt es Argumente, dass ein einmal etabliertes Organell variieren und auch Gene in das Chromosom des Wirtes transportieren kann. Die Erforschung solcher Evolutionsprozesse, deren Reichweite derzeit schwer abzuschätzen ist, bleibt spannend.
|  |
|
Einführung
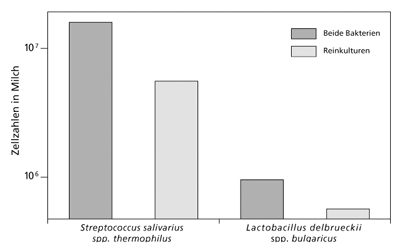 |
| Abb. 1: Metabiose zwischen Streptococcus salivarius spp thermophilus und Lactobacillus delbreuckii spp bulgaricus, den beiden für die Joghurt-Produktion verantwortlichen Milchsäurebakterien. Wenn Milch mit den einzelnen Bakterien beimpft wird, wachsen sie zu deutlich niedrigeren Zellzahlen heran, als wenn beide zusammen in die Milch gegeben werden. Dafür sind vielfältige metabiotische Interaktionen verantwortlich. (Nach Bautista et al. 1966) |
|
Es ist wenig verwunderlich, dass in ökologisch zusammenhängenden Gefügen alle Lebewesen in mehr oder weniger engen Beziehungen zueinander stehen. Die allseits bekannten Nahrungsketten legen davon ein beredtes Zeugnis ab. Darüber hinaus sind jedoch auch viele spezifischere Assoziationen bekannt. Dazu gehören weit verbreitete Metabiosen, bei denen zwei Partner zu gegenseitigem Nutzen zusammenleben. Dabei kann jeder Partner auch alleine existieren, zusammen wachsen sie aber besser. Ein in unserer westlichen Nahrungskultur nahezu allgegenwärtiges Beispiel sind die Joghurt fermentierenden Milchsäurebakterien Lactobacillus delbrueckii ssp bulgaricus und Streptococcus salivarius ssp thermophilus (Abb. 1).
Die negative Variante der Metabiose nennt man Antagonismus. Hier hat nur ein Partner einen Nutzen, der andere wird mehr oder weniger gezielt und nachhaltig geschädigt. Gute Beispiele finden sich bei der Produktion „biologischer Kampfstoffe“ durch Bakterien (z.B. Bakteriozine), durch welche Konkurrenten beseitigt werden (Abb. 2).
Zahlreiche weitere Beispiele finden sich bei bakteriellen Krankheitserregern, die ihren Wirt sogar töten können, jedoch sehr gut ohne Wirt in der Umwelt überleben (Salmonellen oder Listerien). Wenn die gegenseitige Abhängigkeit obligat ist, also ein Partner nicht mehr ohne den anderen leben kann, dann wird die Metabiose zur klassischen Symbiose. Viele Bakterien leben in symbiotischer Beziehung mit ihren Wirten, wir werden in diesem Artikel Beispiele dafür besprechen. Die negative Variante der Symbiose ist der Parasitismus, wobei definitionsgemäß nur der Parasit einen Nutzen hat, nicht jedoch der Wirt. Ein Parasit kann nicht ohne Wirt leben, der Wirt kann aber ohne Parasit auskommen. Bekannte Beispiele für Parasiten unter Bakterien sind obligat parasitäre humane Krankheitserreger wie beispielsweise Mycoplasmen oder Chlamydien. Manchmal ist es gar nicht bekannt, ob eine Beziehung obligat symbiotisch oder obligat parasitisch ist (Abb. 3).
 |
|
Mit * versehene Begriffe
werden im Glossar erklärt.
|
 |
|
Wie alles Leben unterliegen auch diese vielfältigen Beziehungsgefüge zwischen Bakterien und ihren Wirten experimentell zugänglichen mikroevolutiven Prozessen wie Mutations- und Rekombinationsvorgängen verschiedenster Art, Selektion und neutraler Evolution. Auf hohem Niveau sind diese Prozesse beispielsweise in Ridley (2005), allgemeinverständlicher in Junker & Scherer (2006) beschrieben. Leben ist nicht statisch und Organismen verändern sich laufend. Die evolutionsbiologische Forschung hat gezeigt, dass Mikroevolution eines der Grundkennzeichen des Lebens ist. Nukleinsäuresequenzen von Genen können sich ändern, Gene können innerhalb eines Genoms oder auch zwischen Genomen verschiedener Individuen hin und her transportiert werden (horizontaler Gentransfer) und DNA-Sequenzen können ganz verloren gehen (Deletion). Der letztgenannte Vorgang gehört zu den reduktiven Evolutionsprozessen, die bei Symbionten und Parasiten häufig sind.
|
Abb. 2: Antagonismus bei Bakterien. In der Abbildung ist die Wirkung eines Bakteriozins gezeigt. Bakteriozine sind Proteine, die von einem Bakterium ausgeschieden werden und andere Bakterien im Wachstum hemmen. In die ausgestanzten Löcher auf der Agarplatte wurde eine Bakteriozinlösung gefüllt. Die Agarplatte ist mit einem Rasen des Krankheitserregers Listeria monocytogenes bewachsen (trübe Fläche). Rings um Bakteriozin-positive Löcher findet sich eine klare, ringförmige Zone, in die das Bakteriozin hinein diffundierte und das Wachstum des Erregers hemmte. In diesem Experiment handelt es sich um das Bakteriozin Linocin M18, das von dem coryneformen Brevibacterium linens gebildet wird (Valdes-Stauber & Scherer 1994). |
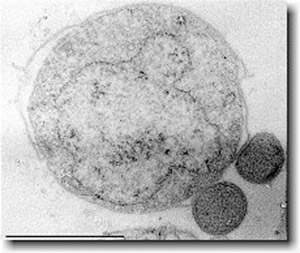 |
| Abb. 3: Das kleinste bekannte Archaebakterium Nanoarchaeum equitans („Reitender Urzwerg“) lebt in obligater Symbiose oder Parasitismus auf anderen Archaebakterien (unten rechts). Sein Genom umfasst nur 490 kb. Es wurde von dem Mikrobiologen Karl O. Stetter entdeckt. Bildrechte: Public domain. |
|
|
|
Intrazelluläre Symbionten von Blattflöhen
 |
| Abb. 4: Blattflöhe (Psyllidae). Foto von John A. Weidhass, Virginia Polytechnical Institute and State University, USA. (www.forestryimages.org/...), Abdruck mit Genehmigung des Autors. |
|
Zahlreiche Insekten leben mit intrazellulären, obligaten Endosymbionten* (Übersicht in Wernegreen 2002 und Baumann et al. 2006). Darunter befinden sich auch die Blattflöhe (Psyllida), vierfügelige Insekten der Unterordnung der Pflanzenläuse (Tab. 1). Die kleinen, nur wenige Millimeter großen Tiere (Abb. 4) besiedeln Pflanzen und ernähren sich von Pflanzensaft, den sie durch Anbohren des Phloems mit einem spezialisierten Bohrrüssel gewinnen. Phloemsaft ist reich an Kohlenhydraten, aber arm an Stickstoffverbindungen (z.B. Aminosäuren). Jede Zelle weist ein relativ konstantes Stickstoff : Kohlenstoff-Verhältnis auf. Wird entweder zu viel C oder zuviel N aufgenommen, muss der jeweilige Überschuss ausgeschieden werden. Um ein ausgewogenes N:C-Verhältnis zu erhalten, scheiden Blattflöhe daher Kohlenhydrate in großen Mengen als klebrige, zuckerreiche Flüssigkeit aus. Diese bedeckt die Pflanzenoberfläche und dient nicht selten als Nahrungsquelle für Pilze. Blattflöhe können massenhaft auftreten und sind als Pflanzenschädlinge gefürchtet.
Seit 1930 ist bekannt, dass Blattflöhe bakterielle Symbionten enthalten, die in einem besonderen Organ (Bacteriom) in der Körperhöhle (Hoemocoel) der Insekten leben (Baumann et al. 2006). Dort leben sie intrazellulär in spezialisierten Zellen. Die Bacterioma befinden sich in nächster Nachbarschaft zum Verdauungstrakt der Insekten. Es handelt sich um eine echte Symbiose, die Bakterien sind ohne ihren Wirt nicht lebensfähig und können daher auch nicht auf künstlichen Wachstumsmedien im bakteriologischen Labor angezüchtet werden, was ihre Untersuchung sehr erschwert. Andererseits können die Insekten nicht ohne ihre bakteriellen Symbionten überleben, welche von den Weibchen über „Infektion“ der Eizellen direkt an die Nachkommen weitergegeben werden. Diese Symbionten bezeichnet man auch als primäre Endosymbionten, im Gegensatz zu weiteren im Hoemocoel lebenden Bakterienarten, auf die der Wirt verzichten kann. Die DNS-Sequenzanalyse von PCR-Amplifikaten* der ribosomalen DNA der primären Endosymbionten ergab, dass es sich um Gram negative* Arten aus der g-Unterabteilung der Proteobakterien handelt. Die primären Symbionten aller untersuchten Psylliden-Gattungen gehören zur Art Carsonella ruddii, wobei die Gattung nur diese eine Art aufweist (Thao et al. 2000). Neben Carsonella leben in Blattflöhen auch noch weitere Endosymbionten (Fukatsu & Nikoh 1998).
|
|
Kleinstes „bakterielles“ Genom
 |
| Tab. 1: Systematische Stellung der Pflanzenläuse und ihrer Unterordnungen. In diesem Artikel geht es um die Blattläuse und Blattflöhe. |
|
Untersuchungen an mehreren Genen von Carsonella ergaben Hinweise auf einen extremen „Minimalismus“: Beispielsweise sind die Gene kürzer als die der nächst ähnlichen, frei lebenden Arten, die Sequenzen zwischen Genen sind stark verkürzt oder gar nicht vorhanden und der GC-Gehalt (das Verhältnis von Guanin zu Cytosin in der DNS) einiger Gene ist mit 10% extrem niedrig (Clark et al. 2001). Letzteres bedeutet, dass teilweise nur 5 Aminosäuren die Hälfte der Aminosäuren eines Proteins stellen. (Zum Vergleich: Durchschnittlich sollten 10 Aminosäuren die Hälfte eines Proteins stellen.) Das sind deutliche Hinweise auf eine enge Symbiose zwischen Bakterium und Insekt und ein möglicherweise kleines Genom des Symbionten. Trotz dieser Hinweise brachte die Genomsequenzierung von Carsonella eine echte Überraschung: Das Genom hat eine Gesamtlänge von nur 159.662 Basenpaaren, also rund 160 kb* (Nakabachi et al. 2006). Zum Vergleich: Das nächst größere bekannte bakterielle Genom ist 422 kb groß und gehört zu einer Buchnera-Art (Perez-Brocal et al. 2006), einem obligaten Symbionten der Blattlaus Cinara cedri. Das Carsonella-Genom enthält gerade einmal 182 Gene. Zahlreiche bisher für unverzichtbar gehaltene Gene fehlen, daher sind beispielsweise weder Lipid- noch Zellwandsynthesen möglich. Die Autoren der Studie stellen fest, dass diese Genausstattung für viele erforderliche bakterielle Stoffwechsel-Abläufe (Metabolismus) eines Bakteriums unzureichend ist. Wie bei allen echten Symbiosen ist der Symbiont also darauf angewiesen, dass verschiedene Stoffe (Metabolite) vom Wirt zur Verfügung gestellt werden.
|
|
Kontinuum der Genomgrößen von Organellen und Bakterien?
Die DNA von Mitochondrien* von Säugetieren hat mit rund 16 kb nur etwa ein Zehntel der Länge des Genoms von Carsonella. Allerdings existieren auch Mitochondrien mit deutlich längerer DNA – die größten Genome von Pflanzenmitochondrien weisen eine Länge von nahezu 500 kb auf (Tab. 2). Die DNA-Größe von Chloroplasten scheint mit 71-200 kb etwas weniger variabel, allerdings ist die verfügbare Datenbasis deutlich geringer als bei Mitochrondrien.
Hinsichtlich der Genomgröße deuten die Daten zunehmend auf ein nahezu geschlossenes Kontinuum hin, welches zwischen Mitochondrien, Chloroplasten und frei lebenden Bakterien vermittelt. Genomgrößen unter 1000 kb (= 1 Mb) wurden bisher ausschließlich bei obligaten Parasiten oder Endosymbionten gefunden. Es sieht so aus, als ob die experimentell beobachtete minimale Genomgröße für ein autonom wachsendes Bakterium etwa in dieser Größenordnung liegt, also bei rund 1000 Genen. Die Übergänge sind bezüglich den Genomgrößen eindeutig fließend, es ist keine klare Größengrenze zwischen frei lebenden Bakterien, parasitisch lebenden Arten wie Mycoplasma genitalium, endosymbiontisch lebenden Arten wie Buchnera und Mitochondrien bzw. Plastiden* auszumachen. Die Genomlänge von Carsonella ergänzt dieses Kontinuum eindrücklich.
|
|
Deutung im Rahmen der Endosymbiontenhypothese
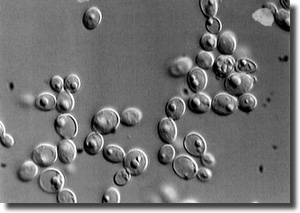 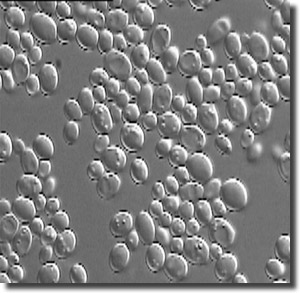 |
Abb. 5: Die beiden Hefen Candida albicans (A) und Candida glabrata (B) haben unterschiedliche Größen ihres mitochondrialen Genoms, vergleiche Tab. 3. Beide Arten sind opportunistische Krankheitserreger. Fotos: Herbert Seiler, Lehrstuhl für Mikrobielle Ökologie, Technische Universität München.
|
|
Zunächst ist festzuhalten, dass sich die genannten, vergleichend biologischen Befunde bezüglich der Genomgrößen gut im Sinne der Endosymbiontentheorie deuten lassen, nach welcher Mitochondrien (und andere DNA-haltige Zellorganellen) sich aus frei lebenden Bakterien durch reduktive Evolution gebildet haben (Rodriguez-Ezpeleta et al. 2005; Cavalier-Smith 2006). Stark divergierende Größen von Chloroplasten- und Mitochrondriengenomen würden dazu passen, in Tab. 2 wird nur eine Auswahl bekannter Daten präsentiert. Außerdem könnte man erwarten, verschiedene Stadien des Genomverlustes bei sehr nahe verwandten Arten zu finden. Dies wird durch die vorliegenden Daten gestützt. Unter anderem ist hier der Vergleich von Genomgrößen von Mitochondrien aus Hefen der Gattung Candida aufschlussreich (Abb. 5).
Sequenzanalysen ergaben Genomgrößen zwischen 20 und 40 kb (Tab. 3). Da diese Hefen einander sehr ähnlich sind, und wahrscheinlich sogar kreuzen, könnte man diesen Befund im Sinne eines erst kurz zurückliegenden Reduktionsvorganges eines mitochondrialen Genoms werten. Allerdings muss noch geklärt werden, welche Gene betroffen sind, welche davon „verschwunden“ sind und welche sich im Kerngenom befinden.
Auch stark unterschiedliche Genomgrößen bei nahe verwandten obligaten endosymbiontischen Bakterien sind bekannt (Wernegreen 2002). Ein gut untersuchtes Beispiel sind unterschiedliche Buchnera aphidicola Stämme. Diese Bakterien leben ebenfalls in Insekten aus der Gruppe der Pflanzenläuse, jedoch aus der Unterordnung der Aphidina
(Blattläuse). Buchnera gehört wie Carsonella zu den g-Proteobakterien*. Aus unterschiedlichen Blattläuse-Gattungen wurden die Buchnera-Endosymbionten sequenziert (für Referenzen siehe Perez-Brocal et al. 2006). Die Endosymbionten haben teilweise deutlich unterschiedliche Genomgrößen (Tab. 3).
 |
|
Yoon et al. (2006) meinen, es sei
„ein Mysterium, wie Organellen
zuerst in Eukaryonten etabliert wurden.“
|
 |
|
Die molekulartaxonomische Analyse ergibt aber ähnliche Stammbäume der Wirte und der Endosymbionten, woraus zunächst auf eine primäre Etablierung der Symbiose vor der Aufspaltung der Aphidina in die vier untersuchten Arten geschlossen wird; der Vorfahr der vier Arten hatte vermutlich schon ein massiv reduziertes Genom, weil die reduzierte Genausstattung der verschiedenen bakteriellen Endosymbionten ziemlich ähnlich ist. Außerdem scheint zwischen den Endosymbionten nach Trennung der Wirte kein signifikanter Genaustausch mehr stattgefunden zu haben, sonst würde man nicht erwarten, dass die molekularen Stammbäume der Endosymbionten und der Wirte übereinstimmen. Dies ist jedoch der Fall.
|
|
Organellen und Endosymbionten
 |
| Tab. 3: Verschiedene Arten der Hefegattung Candida und des Blattlaussymbionten (Buchnera) divergieren in der Größe ihrer mitochondrialen DNA. Dieser Variabilität steht die außerordentliche Konstanz der DNA-Größe von Säugetiermitochondrien gegenüber, die so gut wie gar nicht von 16 kb abweicht. Die Gründe für solche Unterschiede sind nicht genau bekannt. Daten aus http://www.bch. umontreal.ca/ogmp/projects/other/all_list.html, Zugriff am 13. 1. 2007 |
|
Zurück zu Carsonella: Ist Carsonella in Wirklichkeit vielleicht gar kein Bakterium, sondern ein Organell, welches sich im Cytoplasma einer eukaryonten* Zelle eines Insektenbacterioms (Steiner et al. 2002) befindet? Dieses Organell hätte dann ganz andere physiologische Funktionen als Mitochondrien und Chloroplasten. Doch was unterscheidet ein Organell von einem bakteriellen Endosymbionten? Beide sind nicht alleine lebensfähig, und in beiden Fällen existiert ein vitaler Austausch von Metaboliten zwischen den Partnern. Man könnte obligate Endosymbionten und Parasiten mit extrem reduzierter DNA von Organellen vielleicht dadurch unterscheiden, dass bei Organellen Proteine aus dem Cytoplasma des Wirtes in das Organell transportiert werden (Theissen & Martin 2006), wobei zuvor ein funktionaler Gentransfer* in das Genom des Wirtes im Rahmen der Reduktion des Endosymbionten stattgefunden hat. Das ist allerdings ein außerordentlich komplexer Prozess und es ist unklar, wie ein angenommener Transfer der Bakteriengene in das Genom des Wirtes und vor allem der ebenso notwendige Rücktransport der Genprodukte in den Symbionten erstmalig evolviert sein könnte. Yoon et al. (2006) meinen, es sei „ein Mysterium, wie Organellen zuerst in Eukaryonten etabliert wurden.“ Aus diesem Grunde nehmen viele Evolutionsbiologen mit Cavalier-Smith & Lee (1985) an, dass ein solch komplexer, primärer Evolutionsprozess nur zweimal in der Geschichte des Lebens erfolgt sei: Endosymbiontische Bakterien sollen zu Mitochondrien und endosymbiontische Cyanobakterien zu Plastiden evolviert sein (Rodriguez-Ezpeleta et al. 2005). Für die Diskussion der mechanistischen Probleme primärer Endosymbiosen sei der Leser auf Wagner (2006) und Bodyl et al. (2007) verwiesen.
Hier soll diskutiert werden, inwiefern es aus der vergleichenden Biologie inzwischen Argumente für weitere primäre Endosymbiosen gibt. Die Existenz von typischen Organellen, welche eindeutig keine Ähnlichkeit mit Mitochondrien oder Plastiden und vielleicht sogar andere Funktionen haben, könnte in dem Sinne interpretiert werden, dass die unabhängige Bildung von Organellen doch häufiger postuliert werden muss. Allerdings wüsste man damit noch nicht, wie der unbekannte Mechanismus eines derartigen multiplen funktionalen Gentransfers arbeitet. Aus vergleichend-biologischen Daten kann grundsätzlich nicht sicher geschlossen werden, dass ein evolutionärer Mechanismus existiert, der einen vermuteten Evolutionsvorgang in der Vergangenheit ermöglicht haben könnte.
|
|
Carsonella und Paulinella als Beispiele für neuartige Organellen?
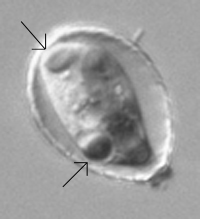 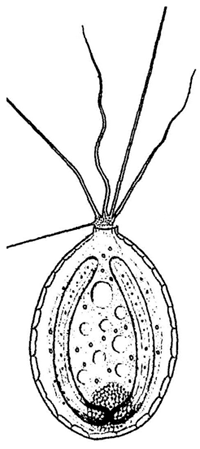 |
Abb. 6: Oben: Paulinella chromatophora, ein Protist in Symbiose mit einem Cyanobakterium (früher: Blaualge). In jeder Zelle liegen zwei „wurstförmige“, DNA-haltige Chromatophoren. Links (zwei Pfeile) ist die Aufsicht auf die kreisformig erschienenden „Enden“ eines Chromatophors im Fokus der Aufnahme. Aufnahme: Y. Tsukii, Japan. Unten: Schemazeichnung von Paulinella des Entdeckers Robert Lauterborn von 1895 (Zeitschr. Wiss. Zool. 59, 537-544).
|
|
Carsonella wäre ein Kandidat für ein neuartiges Organell. Es wird vermutet, dass einige der für das Überleben notwendigen Gene des Endosymbionten in das Genom des Insektes gewandert sind, obgleich es dafür bisher noch keine experimentellen Belege gibt (Nakabachi et al. 2006). Die Genprodukte (Proteine) müssten dann vom Wirtscytoplasma in den Symbionten (zurück)transportiert werden. Man könnte diese Hypothese für Carsonella relativ leicht prüfen, indem das Proteom (Gesamtheit aller Proteine) des Endosymbionten analysiert wird. Dabei würde man die gesamte Proteinausstattung isolierter Bacteriome im zweidimensionalen elektrophoretischen Gel auftrennen, die Proteine massenspektroskopisch identifizieren und dann durch Vergleich sehen, ob alle Carsonella-Proteine auch auf dem Carsonella-Genom codiert sind. „Fremde“ Proteine müssten aus dem Wirt importiert worden sein. Ein weiterer Schritt bestünde in der Sequenzanalyse der gefundenen fremden Gene aus dem Genom des Blattflohs und dem Vergleich mit den nächst ähnlichen Sequenzen. Wenn diese bei nahen frei lebenden Verwandten von Carsonella (also Arten aus der g-Unterabteilung der Proteobakterien) vorhanden wären, dann könnte man die Daten auf einer vergleichenden Ebene sehr gut im Sinne eines weitgehend fortgeschrittenen Endosymbiosevorganges in Richtung eines neuartigen Organells deuten.
Ein zweiter, derzeit intensiv diskutierter Kandidat für eine neue, primäre Organellenbildung ist die Amöbe Paulinella chromatophora. Seit vielen Jahrzehnten ist bekannt, dass diese Amöbe mit einem intrazellulären Cyanobakterium obligat vergesellschaftet ist (Abb. 6).
Der Endosymbiont wird wegen der Präsenz einer Zellwand aus Peptidoglykan und den typischen photosynthetischen Pigmenten von Cyanobakterien (Phycobilisomen) auch als Cyanelle bezeichnet. Der Endosymbiont liegt frei im Cytoplasma der Wirtsamöbe, ist also nicht von
 |
|
Aus vergleichend-biologischen Daten
kann grundsätzlich nicht sicher
geschlossen werden, dass ein evolutionärer
Mechanismus existiert, der einen
vermuteten Evolutionsvorgang in der
Vergangenheit ermöglicht haben könnte.
|
 |
|
einer Phagocytosemembran der Wirtszelle umgeben. Er kann nicht alleine kultiviert werden und die Zahl der Endosymbionten wird von der Wirtszelle auf unbekannte Weise reguliert. Damit ist die Cyanelle von Paulinella kein einfacher „Kleptoplast“, der immer wieder durch Phagocytose* aufgenommen wird und nach einigen Verdopplungen wieder verloren geht. Dieser Endosymbiont ist genetisch sehr verschieden von den echten Organellen der Glaucocystophyta (Marin et al. 2005), die Phycobilisomen und Zellwandreste tragen und nächstverwandt mit Chloroplasten und Rhodoplasten sind (Rodriguez-Ezpeleta et al. 2005). Die Cyanelle von Paulinella steht nach der rDNA*-Sequenz demgegenüber eindeutig einem Cyanobakterium aus der Synechococcus-Gruppe nahe. Die Ergebnisse von Marin et al. (2005) wurden daher als wichtiger Befund für eine dritte primäre Endosymbiose gewertet (Archibald 2006; Rodriguez-Ezpeleta & Philippe 2006; Bodyl et al. 2007). Weitere genetische Analysen der Cyanelle zeigten jedoch, dass es sich nicht um ein Organellengenom, sondern um ein Cyanobakterien-Genom handelt (Yoon et al. 2006; Marin et al. 2007). Ob dieser Endosymbiont ein teilweise reduziertes Genom besitzt, wurde noch nicht generell untersucht, die bisher bekannten DNA-Sequenzen sprechen aber nicht dafür.
|
|
Endosymbiose in statu nascendi?
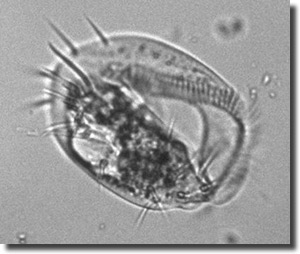 |
| Abb. 7: Euplotes sp. gehört zu den Ciliophora. Der Protist ist etwa zwischen 0,05 und 0,1 mm lang und kann Bakterien als Endosymbionten beherbergen (vgl. Text). Aufnahme: H. Yoshino, Japan. |
|
Die besprochenen Endosymbiosen weisen meist extreme Genomreduktionen der Mikrosymbionten auf. Das muss aber nicht immer so sein. Am anderen Ende der Skala stehen obligate Endosymbionten, die sich kaum von frei lebenden, nächstverwandten Isolaten der gleichen Art unterscheiden. Anfang 2007 haben Vannini et al. (2007) erstmalig ein solches Beispiel beschrieben. Es handelt sich um Polynucleobacter necessarius, ein Mitglied der b-Proteobakterien. Diese Art lebt sowohl als obligater Endosymbiont in dem Ciliaten Euplotes, aber auch freilebend im Pelagial* von Süßwasserhabitaten. Euplotes (Abb. 7) kann nicht ohne den Symbionten leben und endosymbiontisch lebende Polynucleobacter sind nicht kultivierbar.
40 freilebende und 5 endosymbiotische Stämme wurden verglichen. Sie unterschieden sich nur in 1,5% ihrer 16S rDNA-Sequenzen und gehören damit sehr wahrscheinlich zur gleichen Spezies. Die Isolate stammen aus Südamerika, Australien, China und Europa. Auffallenderweise gruppierten die endosymbiontischen Isolate nach ihren Wirten und nicht mit ihren frei lebenden Speciesgenossen. Außerdem scheinen die endosymbiontisch lebenden Polynucleobacter mit 1700 bis 1800 kb etwas kleinere Genome aufzuweisen als die pelagischen Arten der gleichen Ähnlichkeitsgruppe (2100 bis 2500 kb), obgleich es auch einzelne pelagische Arten mit kleineren Genomen gibt. Diese Daten sprechen bisher dafür, dass Vannini et al. eine initiale Form der obligaten Endosymbiose gefunden haben. Interessanterweise scheint diese Assoziation spezifisch zu sein, d.h. sowohl Euplotes als auch Polynucleobacter können nicht mit beliebigen anderen Partnern eine Symbiose eingehen. Welche Grundlage diese Prädisposition hat, ist im Einzelnen unbekannt.
|
|
Fähigkeit zur Symbiose als Kennzeichen des Lebens?
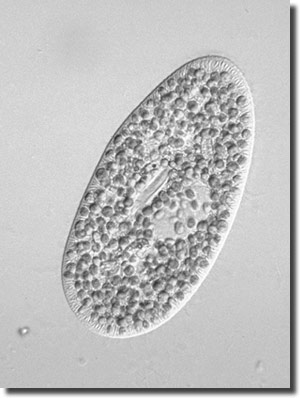 |
| Abb. 8: Dieser Vertreter der Pantoffeltierchen (Paramecium bursaria) ist angefüllt mit Grünalgen der Art Chlorella. Diese versorgen den Wirt mit Photosynheseprodukten, der Wirt scheint für Schutz und Transport des Endosymbionten zu sorgen. Foto: Y. Tsukii, Japan. |
|
Vieles spricht dafür, dass Assoziationen von Mikroorganismen mit Wirten aller Art viel häufiger sind als bisher angenommen wurde. Auch für bisher „unverdächtige“ Bakterien mehren sich Hinweise auf symbiotische und/oder parasitische Beziehungen, besonders zu Wirten von Wirbellosen und eukaryonten mikrobiellen Wirten. So konnten wir kürzlich zeigen, dass der bisher nur aus Umweltisolaten bekannte humane Krankheitserreger Yersinia enterocolitica insektentoxische Proteine bildet (Bresolin et al. 2006), doch der vermutete Insektenwirt ist nicht bekannt. Solche Interaktionen erzeugen neue ökologische Nischen und damit interessante Evolutionsprozesse.
Vielleicht gehört es sogar zur genetischen Grundausstattung von mindestens einigen, vielleicht sogar vielen Bakteriengruppen, enge Verbindungen zu verschiedensten Wirten eingehen zu können. Allerdings müssten auch die potentiellen Wirte genetisch dafür prädisponiert sein. Einige wenige Beispiele seien genannt: Methanbildende Bakterien leben in großer Vielfalt in anaeroben Ciliaten und zeigen „ausgeklügelte morphologische Interaktionen“ (Embley & Finlay 1993), die vermutlich den Stoffaustausch zwischen den Partnern erleichtern. Viele Grünalgen leben intrazellulär vergesellschaftet mit Ciliaten, z. B. dem Pantoffeltierchen (Abb. 8).
Bei einer Kieselalge wurde ein intrazellulär lebendes Cyanobakterium gefunden, welches keine Zellwand und keine Photosynthese aufweist, obligat endosymbiontisch lebt und vermutlich als stickstofffixierender Endsymbiont dient (Prechtl et al. 2004). Überhaupt leben viele stickstofffixierende Bakterien eng mit Insekten, Schnecken, Pilzen oder Pflanzen zusammen (Thacker 2005; Vega et al. 2006; Kneip et al. 2007). Es gibt erste Schätzungen, dass mindestens 10% aller Insektenspezies mit obligaten (!) Endosymbionten leben (Dale & Moran, 2006). Kein Zweifel besteht daran, dass alle Organismen mit einem Magen-Darm-Trakt in enger Verbindung mit zahlreichen, in der Regel unbekannten Mikroben leben, wobei die funktionalen Interaktionen weitgehend unbekannt sind. Das gilt auch für den Menschen (Ley et al. 2006; Turnbaugh et al. 2006), in dessen Darmtrakt ca. 1013 Bakterien mit geschätzten 1000 Arten leben.
|
|
Variationspotential durch Gentransfer?
Welche Variabilität ist möglich, wenn die Gen- und Proteintransfermechanismen in einem komplexen System mit Mitochondrien und/oder Plastiden erst einmal vorhanden sind? Ist in einem solchen kompletten System der (funktionale?) Transfer weiterer Gene in den Zellkern der Wirtszelle wahrscheinlich? Die in Tab. 2 dargestellten Variationen der Genomgrößen von Hefen könnten ein Indiz dafür sein. Auch einige an Pflanzenorganellen (Adams et al. 1999; Palmer et al. 2000; Adams et al. 2000) erhobene Befunde sprechen dafür. Für Mitochondrien von Hefen gibt es inzwischen experimentelle Daten, dass ein Gentransfer vom Mitochondrium in den Kern mit anschließender Transkription (Abschrift) des Gens im Kern und Translation (Übersetzung in die Abfolge der Aminosäuren eines Proteins) im Cytoplasma unter geeigneten Bedingungen im Labor beobachtet werden kann (Thorsness & Fox 1990; Campbell & Thorsness 1998; Ricchetti et al. 1999). Vergleichend-biologische Befunde an Pflanzen wurden so gedeutet, dass dieser Prozess sogar überraschend schnell abläuft (Gray 2000). Stellen Organellen die funktionalen Mechanismen bereit, die im Rahmen zusätzlicher Endosymbiosevorgänge genutzt werden können? Bodyl et al. (2007) vermuten, dass „die anfängliche Etablierung des Mitochondriums eukaryonte Zellen für weitere endosymbiontische bakterielle Assoziationen präadaptiert haben könnte.“
Möglicherweise haben wir erst damit begonnen, die Vielfalt der in Lebewesen realisierten Variationsmechanismen zu verstehen, die mit einer auf Symbiose bzw. Parasitismus programmierten Lebensweise einhergehen könnten. Kein Zweifel: Die Erforschung dieser Variationsprozesse im Rahmen der Evolutionsbiologie wird auf absehbare Zeit spannend bleiben.
|
Glossar
- Endosymbiont:
- Ein im Zellinneren eines Wirtes lebender Organismus
- Eukaryont:
- Zelle mit Zellkern
- Genom:
- Das komplette Erbgut eines Organismus
- Gentransfer:
- Übertragung von Genen von einem Organismus in einen anderen oder von einem Organell oder Endosymbionten in den Zellkern
- Gram negativ:
- Gram negative Bakterien sind nach einer bestimmen Färbemethode, die vom Bakteriologen Hans Christian Gram entwickelt wurde, farblos. Nach dieser Färbemethode werden Bakterien in Gram positive und Gram negative Arten unterteilt. Der Unterschied in der Färbung ist durch Zellwandeigenschaften bedingt.
- kb:
- Kilobase = 1000 Basen. „Basen“ ist die Kurzbezeichnung für die Nucleobasen Thymin, Adenin, Guanin und Cytosin, aus denen die DNA kettenförmig zusammengesetzt ist.
- Mitochondrium:
- Membranumschlossenes Zellorganell, das als „Kraftwerk“ der Zelle fungiert und eine eigene DNA besitzt
- PCR-Amplifikat:
- Mittels „polymerase chain reaction“-Methode vervielfältigte DNA
- Pelagial:
- ufernaher Freiwasserbereich in Seen und im Meer.
- Phagocytose:
- Aufnahme von größeren Partikeln in Zellen.
- Phloem:
- Leitgewebe zum Transport von Nährstoffen in Pflanzen
- Plastid:
- Membranumschlossene Zellorganelle pflanzlicher Zellen. Beispielsweise findet in den Chloroplasten die Photosynthese statt. Auch die Plastiden besitzen eine eigene DNA.
- Proteobakterien:
- Proteobakterien gehören zu den Gram negativen Bakterien. Zu dieser Untergruppe gehören auch Enterobakterien wie z. B. Escherichia coli und Salmonella.
- rDNA:
- Diese DNA kodiert für die ribosomale RNA, welche Bestandteil der Ribosomen (Ort der Proteinsynthese) ist.
|
Literatur
- Adams KL, Daley DO, Qiu YL, Whelan J & Palmer JD (2000)
- Repeated, recent and diverse transfers of a mitochondrial gene to the nucleus in flowering plants. Nature 408, 354-357.
- Adams KL, Song K, Roessler PG, Nugent JM, Doyle JL, Doyle
- JJ & Palmer JD (1999) Intracellular gene transfer in action: dual transcription and multiple silencings of nuclear and mitochondrial cox2 genes in legumes. Proc. Natl. Acad. Sci. USA 96, 13863-13868.
- Archibald JM (2006)
- Endosymbiosis: double-take on plastid origins. Curr. Biol. 16, R690-692.
- Baumann P, Moran NA & Baumann L (2006)
- Bacteriocyte-Associated Endosymbionts of Insects. In The Prokaryotes. Dworkin M (ed) New York: Spinger, pp. 403-438, DOI 410.1007/1000-1387-30741-30749_30716.
- Bautista ES, Dahiya RS & Speck ML (1966)
- Identification of compounds causing symbiotic growth of Streptococcus thermophilus and Lactobacillus bulgaricus. J. Dairy Res. 33, 299-307.
- Bodyl A, Mackiewicz P & Stiller JW (2007)
- The intracellular cyanobacteria of Paulinella chromatophora: endosymbionts or organelles? Trends Microbiol. 15, 295-296.
- Bresolin G, Morgan JA, Ilgen D, Scherer S & Fuchs TM (2006)
- Low temperature-induced insecticidal activity of Yersinia enterocolitica. Mol. Microbiol. 59, 503-512.
- Campbell CL & Thorsness PE (1998)
- Escape of mitochondrial DNA to the nucleus in yme1 yeast is mediated by vacuolar-dependent turnover of abnormal mitochondrial compartments. J. Cell. Sci. 111 ( Pt 16), 2455-2464.
- Cavalier-Smith T (2006)
- Origin of mitochondria by intracellular enslavement of a photosynthetic purple bacterium. Proc. Biol. Sci. 273, 1943-1952.
- Cavalier-Smith T & Lee J (1985)
- Protozoa as hosts for endosymbioses and the conversion of symbionts into organelles. J. Protozool. 32, 376-379.
- Clark MA, Baumann L, Thao ML, Moran NA & Baumann P (2001)
- Degenerative minimalism in the genome of a psyllid endosymbiont. J. Bacteriol. 183, 1853-1861.
- Dale C & Moran NA (2006)
- Molecular interactions between bacterial symbionts and their hosts. Cell 126, 453-465.
- Embley TM & Finlay BJ (1993)
- Systematic and morphological diversity of endosymbiotic methanogens in anaerobic ciliates. Antonie Van Leeuwenhoek 64, 261-271.
- Fukatsu T & Nikoh N (1998)
- Two intracellular symbiotic bacteria from the mulberry psyllid Anomoneura mori (Insecta, Homoptera). Appl. Environ. Microbiol. 64, 3599-3606.
- Gray MW (2000)
- Mitochondrial genes on the move. Nature 408, 302-303, 305.
- Junker R & Scherer S (2006)
- Evolution – ein kritisches Lehrbuch. 6. Aufl., Gießen.
- Kneip C, Lockhart P, Voss C & Maier UG (2007)
- Nitrogen fixation in eukaryotes—new models for symbiosis. BMC Evol. Biol. 7, 55.
- Ley RE, Turnbaugh PJ, Klein S & Gordon JI (2006)
- Microbial ecology: human gut microbes associated with obesity. Nature 444, 1022-1023.
- Marin B, Nowack EC & Melkonian M (2005)
- A plastid in the making: evidence for a second primary endosymbiosis. Protist 156, 425-432.
- Marin B, Nowack EC, Glockner G & Melkonian M (2007)
- The ancestor of the Paulinella chromatophore obtained a carboxysomal operon by horizontal gene transfer from a Nitrococcus-like gamma-proteobacterium. BMC Evol. Biol. 7, 85.
- Nakabachi A, Yamashita A, Toh H, Ishikawa H, Dunbar HE, Moran NA & Hattori M (2006)
- The 160-kilobase genome of the bacterial endosymbiont Carsonella. Science 314, 267.
- Palmer JD, Adams KL, Cho Y, Parkinson CL, Qiu YL & Song K (2000)
- Dynamic evolution of plant mitochondrial genomes: mobile genes and introns and highly variable mutation rates. Proc. Natl. Acad. Sci. USA 97, 6960-6966.
- Perez-Brocal V, Gil R, Ramos S, Lamelas A, Postigo M, Michelena JM et al. (2006)
- A small microbial genome: the end of a long symbiotic relationship? Science 314, 312-313.
- Prechtl J, Kneip C, Lockhart P, Wenderoth K & Maier UG (2004)
- Intracellular spheroid bodies of Rhopalodia gibba have nitrogen-fixing apparatus of cyanobacterial origin. Mol. Biol. Evol. 21, 1477-1481.
- Ricchetti M, Fairhead C& Dujon B (1999)
- Mitochondrial DNA repairs double-strand breaks in yeast chromosomes. Nature 402, 96-100.
- Ridley M (2005)
- Evolution. Oxford.
- Rodriguez-Ezpeleta N & Philippe H (2006)
- Plastid origin: replaying the tape. Curr. Biol. 16, R53-56.
- Rodriguez-Ezpeleta N, Brinkmann H, Burey SC, Roure B, Burger G, Loffelhardt W et al. (2005)
- Monophyly of primary photosynthetic eukaryotes: green plants, red algae, and glaucophytes. Curr. Biol. 15, 1325-1330.
- Steiner J, Pfanzagl B, Ma Y & Löffelhardt W. (2002)
- Evolution and biology of cyanelles. Biology and Environment: Proc. Roy. Irish Acad. 102B, 7-9.
- Thacker RW (2005)
- Impacts of Shading on Sponge-Cyanobacteria Symbioses: A Comparison between Host-Specific and Generalist Associations. Integr. Comp. Biol. 45, 369-376.
- Thao ML, Moran NA, Abbot P, Brennan EB, Burckhardt DH & Baumann P (2000)
- Cospeciation of psyllids and their primary prokaryotic endosymbionts. Appl. Environ. Microbiol. 66, 2898-2905.
- Theissen U & Martin W (2006)
- The difference between organelles and endosymbionts. Curr. Biol. 16, R1016-R1017
- Thorsness PE & Fox TD (1990)
- Escape of DNA from mitochondria to the nucleus in Saccharomyces cerevisiae. Nature 346, 376-379.
- Turnbaugh PJ, Ley RE, Mahowald MA, Magrini V, Mardis ER & Gordon JI (2006)
- An obesity-associated gut microbiome with increased capacity for energy harvest. Nature 444, 1027-1031.
- Valdes-Stauber N & Scherer S (1994)
- Isolation and characterization of Linocin M18, a bacteriocin produced by Brevibacterium linens. Appl. Environ. Microbiol. 60, 3809-3814.
- Vannini C, Pockl M, Petroni G, Wu QL, Lang E, Stackebrandt E et al. (2007)
- Endosymbiosis in statu nascendi: close phylogenetic relationship between obligately endosymbiotic and obligately free-living Polynucleobacter strains (Betaproteobacteria). Environ. Microbiol. 9,347-359.
- Vega IA, Damborenea MC, Gamarra-Luques C, Koch E, Cueto JA & Castro-Vazquez A (2006)
- Facultative and obligate symbiotic associations of Pomacea canaliculata (Caenogastropoda, Ampullariidae). Biocell 30, 367-375.
- Wagner G (2006)
- Endosymbionten-Hypothese. http://www.genesisnet.info/schoepfung_evolution/e41308.php
- Wernegreen JJ (2002)
- Genome evolution in bacterial endosymbionts of insects. Nat. Rev. Genet. 3, 850-861.
- Yoon HS, Reyes-Prieto A, Melkonian M & Bhattacharya D (2006)
- Minimal plastid genome evolution in the Paulinella endosymbiont. Curr. Biol. 16, R670-672.
|
|  |
